论文解读| Yang Li/Linhai Li教授团队揭示揭示胃癌预后预测新方法
15小时前 Genes and Diseases Genes and Diseases 发表于陕西省
该研究提出了一种基于AI和单碱基替换(single base substitution, SBS)特征的新型胃癌预后预测模型,为个体化诊疗提供了新的思路。
胃癌作为全球范围内常见的恶性肿瘤之一,其五年生存率的准确预测对于制定有效的临床治疗计划极为关键。然而,传统的肿瘤分期方法主要依赖于统计数据和经验性研究,这限制了它们在提供个体化精确预后信息方面的能力。随着人工智能(AI)技术的发展为胃癌五年生存率的预测提供了新的可能。
广州医科大学的Yang Li/Linhai Li教授团队在本刊发表了题为“AI-driven analysis establishes the single base substitution signatures as personalized prognostic predictors for five-year survival of gastric cancer”的研究快讯,提出了一种基于AI和单碱基替换(single base substitution, SBS)特征的新型胃癌预后预测模型,为个体化诊疗提供了新的思路。
1 研究方法
研究团队收集了来自以往基因组研究的462名胃癌患者的体细胞突变资料,运用基因累积贡献丰度(CCA)模型和随机森林算法,构建了结合SBS44和SBS18特征的胃癌5年生存率预测模型。
2 研究结果
在胃癌样本中,C > T(占比30.14%)和T > C(占比23.44%)突变类型最为常见(图1A)。通过独立的特征提取分析,研究团队进一步确认了SBS44*在胃癌中的显著作用,这一特征与DNA错配修复缺陷紧密相关,并在多个独立样本队列中得到了一致的发现(图1B)。Kaplan-Meier生存分析结果表明,SBS44突变暴露程度超过20%的肿瘤患者,其生存结果显著更佳;相对地,SBS18突变暴露程度超过20%的患者则面临较差的预后(图1D、E)。研究团队构建了一个结合SBS44和SBS18签名的胃癌五年生存率预测AI模型,该模型准确性较高, AUC值为0.9194,95%置信区间介于0.8357至1之间(图1F–I)。
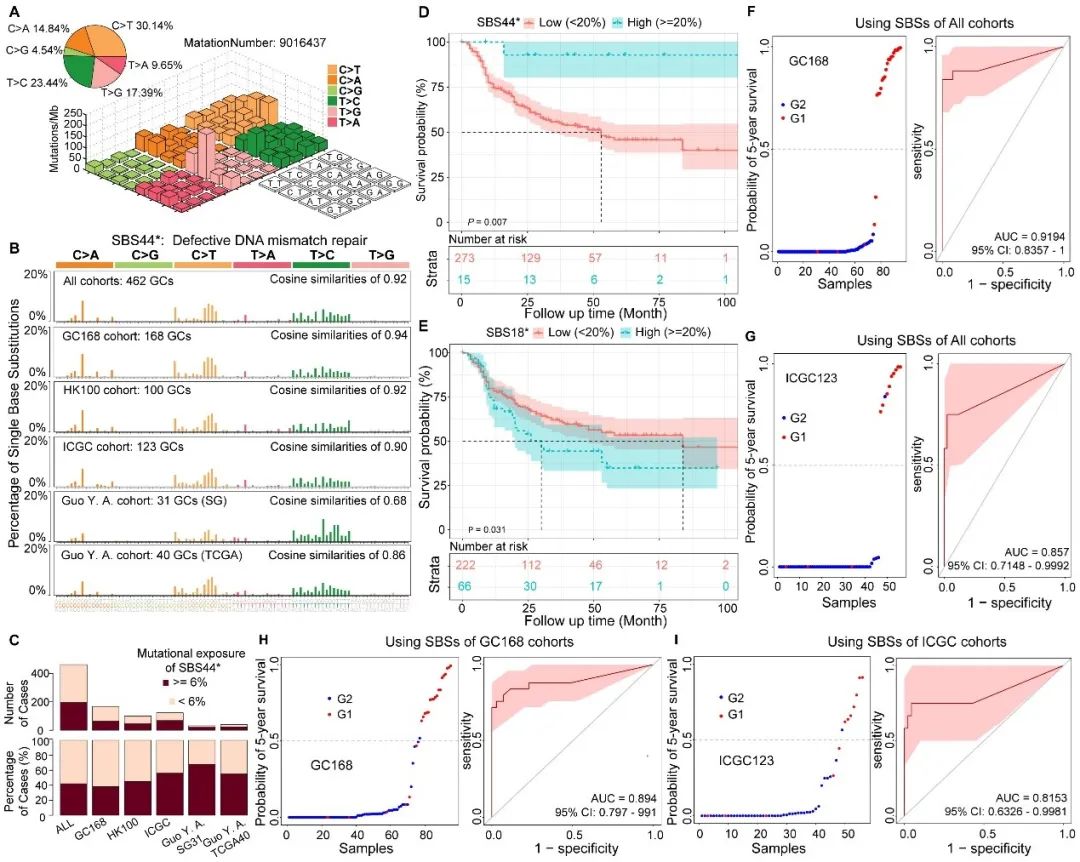
图1 SBS44*与胃癌中单碱基替换(SBSs)的预后
3 研究结论
本研究开发的基于基因与预后相关突变签名的预测模型,能够有效提升胃癌患者预后评估的准确性,对指导临床治疗决策具有显著的辅助作用。这项技术不仅开辟了胃癌预后评估的新途径,其原理和方法亦有望推广至其他类型肿瘤的预后评估中,为个体化医疗提供更为精准的工具。
本文由“壹伴编辑器”提供技术支持
文章来源
免费全文下载链接:
https://www.sciencedirect.com/science/article/pii/S2352304223002982
引用这篇文章:
Li Z, Xu L, Luo W, et al. AI-driven analysis establishes the single base substitution signatures as personalized prognostic predictors for five-year survival of gastric cancer. Genes Dis. 2024;11(4):101030.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言
















#胃癌# #个体化诊疗#
3