Nature Biotechnology:突破细胞类型限制:NIS-Seq开启基因筛选新纪元
4小时前 生物探索 生物探索 发表于陕西省
本文介绍核内原位测序(NIS-Seq)技术,包括其克服传统基因扰动筛选局限、原理、在多种细胞及人皮肤组织中的验证、应用于炎症通路等研究,探讨其优势、局限性与未来发展。
引言
在探索基因与表型之间复杂关系的研究中,基因组水平的扰动筛选(perturbation screening)已成为解锁生物学奥秘的重要工具。然而,传统基于富集策略的筛选方法存在显著的局限性,例如对细胞转录活性、胞质体积和低细胞密度的依赖,这使得它们难以在某些生物学环境中有效实施,尤其是在原代细胞或低转录活性的细胞类型中。为了突破这些瓶颈,研究人员开发了一种创新技术——核内原位测序(Nuclear In-Situ Sequencing, NIS-Seq),其显著特点在于通过在细胞核内生成亮度极高的测序信号,实现了细胞类型不可知的高密度光学筛选。(12月19日 Nature Biotechnology “NIS-Seq enables cell-type-agnostic optical perturbation screening”)
该研究的亮点在于,NIS-Seq技术不仅克服了传统方法对细胞特性的依赖,还能在各种细胞类型中生成一致且清晰的信号。这一技术通过引入T7启动子的反向转录步骤,从基因组DNA中生成大量RNA拷贝,结合锁环延伸(padlock elongation)、滚环扩增(rolling circle amplification)和多轮合成测序,实现了高效、精准的基因扰动识别。研究团队在包括HeLa细胞、THP1来源的巨噬细胞和初级人巨噬细胞在内的八种细胞类型中对NIS-Seq进行了验证,结果显示其在高密度细胞筛选和复杂库映射方面表现出极高的准确性和适用性。
此外,该技术不仅被应用于解码炎症相关通路的关键基因,还成功完成了在原代人巨噬细胞中的小规模筛选,甚至实现了在人皮肤组织中的条形码识别。这些突破展示了NIS-Seq在基础研究和临床转化中的广阔前景,例如细胞迁移、蛋白质复合物形成、动态信号转导等复杂生物过程的研究。

基因扰动筛选的新时代:从传统到创新
在生命科学研究中,基因扰动筛选(perturbation screening)被视为探索基因功能和表型联系的关键工具。这种方法通过随机扰动细胞中的基因组,筛选出与特定生物学现象相关的基因,帮助研究人员解码复杂的遗传网络。然而,传统的基因筛选方法,尽管在推动基因组研究方面取得了显著进展,却仍然面临诸多挑战。
传统方法大多依赖富集策略,例如通过荧光标记或细胞分选技术(如FACS)对特定表型的细胞进行筛选。随后,这些细胞中的基因扰动因子(如CRISPR单链向导RNA, sgRNA)会通过下一代测序(NGS)进行识别。然而,这些方法的效率高度依赖于细胞的转录活性和胞质体积。在低转录活性的细胞类型(如原代细胞)中,信号可能不足,难以进行精确分析。此外,传统方法通常需要将细胞分离到单独的孔或空间中进行测定,这种操作繁琐且容易引入实验偏差。同时,在高细胞密度的环境中,准确定位基因扰动的来源也极具挑战性。
NIS-Seq技术如何颠覆基因组筛选?
核内原位测序(NIS-Seq)是一项突破性技术,它从根本上颠覆了传统基因组筛选的方式,以全新的角度解决了多年来存在的技术瓶颈。
NIS-Seq的核心原理围绕在细胞核内生成明亮且稳定的信号。传统的原位测序依赖于细胞胞质中的信使RNA(mRNA),然而mRNA的表达水平高度依赖细胞的转录活性,在低活性或原代细胞中往往无法生成足够的信号。而NIS-Seq则将信号生成的过程转移到了细胞核内,其关键在于引入了T7反向转录系统。研究人员在单链向导RNA(sgRNA)表达盒中添加了反向的T7启动子,从而使大量sgRNA的RNA拷贝能够直接从基因组DNA转录生成。接下来,这些RNA拷贝通过逆转录形成cDNA,并进一步通过锁环延伸(padlock elongation)、连接(ligation)和滚环扩增(rolling circle amplification, RCA)生成显著增强的信号。
NIS-Seq的另一独特之处在于其多轮荧光测序步骤。通过三色激光的合成测序,每轮测序生成的信号能够明确地标记单细胞核内的基因扰动条形码。这种方法不仅在单细胞水平上提供了极高的分辨率,还避免了传统方法在高细胞密度环境中信号分配模糊的问题。例如,在实验中,研究人员对THP1来源的巨噬细胞进行了14轮测序,生成了精准且一致的核内荧光信号,为基因扰动的高效识别奠定了基础。
通过这些技术创新,NIS-Seq显著提高了基因组筛选的效率和广泛适配性。它突破了传统技术对细胞类型和转录活性的限制,使得在低活性细胞和原代细胞中也能稳定生成清晰信号,为探索基因功能开辟了全新路径。
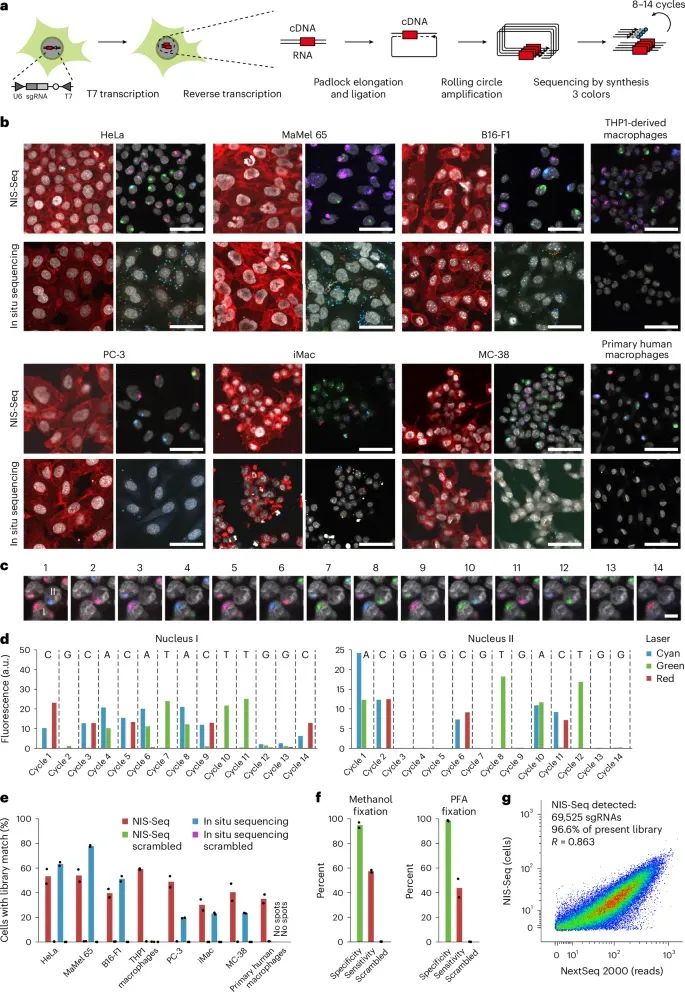
NIS-Seq技术在核内识别基因条形码的能力(Credit: Nature Biotechnology)
a. NIS-Seq反应步骤概述
图a总结了NIS-Seq技术的工作流程。NIS-Seq在传统基于mRNA的原位测序方法基础上,增加了一个T7启动子的转录步骤,直接在细胞核内生成大量信号。这一设计绕过了对细胞转录活性和胞质体积的依赖,为多种细胞类型的适配奠定了基础。
b. 第一轮NIS-Seq成像结果与传统方法对比
图b比较了NIS-Seq与传统细胞胞质原位测序方法在八种细胞类型中的成像效果。结果显示,NIS-Seq在所有细胞类型(包括THP1来源的巨噬细胞和初级人巨噬细胞)中都能生成清晰的核内信号,而传统方法在某些细胞类型中未能产生足够的信号。这表明NIS-Seq在低转录活性和多样化细胞类型中的显著优势。
c. 14轮NIS-Seq测序的原始图像
图c展示了THP1细胞经过14轮NIS-Seq循环测序后的原始图像。实验中在第1、4、7、10和13轮进行了核染色,结果显示每轮测序的核内荧光信号清晰且稳定,表明NIS-Seq能够在多轮测序中保持高分辨率和可靠性。
d. 定量分析核内信号强度
图d对THP1细胞的两个核(Nucleus I和Nucleus II)进行了定量荧光信号强度分析。信号强度和碱基读取结果与条形码序列库中的两个条形码匹配,验证了NIS-Seq在条形码识别上的精准性。
e. 细胞条形码映射率
图e分析了细胞映射到已知条形码库成员的比例。结果表明,在使用NIS-Seq时,高达96.6%的细胞条形码能够被精确映射,而对照组(使用随机排列条形码参考库)中几乎没有信号。这表明NIS-Seq在条形码匹配上的高特异性。
f. NIS-Seq的特异性和灵敏度
图f通过混合细胞实验(表达GFP或NIS-Seq条形码的细胞)评估了NIS-Seq的特异性和灵敏度。实验对比了标准NIS-Seq方法和PFA固定后的改进版,结果显示NIS-Seq在两种固定方法下均能保持极高的特异性和灵敏度。
g. NIS-Seq的基因文库覆盖率
图g将NIS-Seq检测到的基因条形码与基于PCR的下一代测序(NGS)结果进行对比。结果显示,两种方法在基因库覆盖率上具有高度一致性,且相关性高达0.863,进一步验证了NIS-Seq的准确性和可靠性。
八种细胞类型的实验验证:一个技术适配性的突破
NIS-Seq技术的适配性和普适性在基因组筛选领域展现了令人瞩目的潜力。研究人员通过对八种不同类型的细胞进行实验验证,成功证明了NIS-Seq在多种生物学环境中的高效表现。这些细胞类型包括经典的实验室细胞系(如HeLa细胞)、肿瘤细胞(MaMel 65、B16-F1)、免疫细胞(THP1来源的巨噬细胞)以及极具挑战性的原代人巨噬细胞。结果表明,NIS-Seq在所有这些细胞中均能生成亮度极高且分辨率清晰的核内信号,无论细胞的转录活性或形态特性如何。
尤其值得一提的是,NIS-Seq在低转录活性细胞中的表现令人耳目一新。传统的基因组筛选方法在处理这些细胞时往往难以生成足够信号,导致筛选结果受限。而NIS-Seq通过直接在细胞核内对sgRNA的序列条形码进行转录、扩增和测序,有效绕过了对细胞转录活性的依赖。例如,在THP1来源的巨噬细胞和初级人巨噬细胞中,传统的原位测序几乎无法生成清晰的信号,而NIS-Seq却能够产生亮度显著增强的核内信号。此外,即使在高细胞密度的环境中,NIS-Seq依然能实现精准的细胞核映射,使得不同条形码能够被可靠地分配到正确的细胞。
数据进一步验证了这一技术的广泛适用性。研究表明,在所有测试的细胞类型中,NIS-Seq的条形码匹配率和灵敏度均显著高于传统方法。实验中,96.6%的已知序列库条形码能够通过NIS-Seq检测到,而传统原位测序在某些细胞类型中甚至完全丧失信号。这种技术的稳定性和普适性使得研究人员可以在从肿瘤细胞到原代免疫细胞的广泛环境中实施基因组筛选。
通过这次跨越不同细胞类型的实验验证,NIS-Seq不仅展现了强大的技术适配性,更为研究复杂的细胞系统、低转录活性细胞和原代细胞提供了一种高效、精准的工具。
高密度筛选的利器:NIS-Seq的精准度与可靠性
在基因组筛选中,高细胞密度实验一直是技术的试金石——如何在有限空间内精准分配和解读基因条形码信号,直接决定了筛选的成功与否。NIS-Seq凭借其独特的核内信号生成机制和高灵敏度,成为应对这一挑战的“利器”,在高密度细胞环境中展现了前所未有的精准度与可靠性。
传统的筛选方法在高密度条件下容易出现信号交叉或分配错误,因为这些方法依赖于胞质信号,而在密集的细胞环境中,胞质边界常常模糊不清。然而,NIS-Seq通过在细胞核内生成强荧光信号,完全规避了这一问题。在实验中,研究人员利用NIS-Seq对THP1来源的巨噬细胞和其他类型细胞进行了高密度筛选测试,成功地在单核分辨率下映射了多轮测序产生的基因条形码。这一技术能够在核内信号的位置和颜色上做到精确匹配,即使在复杂的细胞排列中也能准确区分每个细胞的基因扰动条形码。
实验数据进一步凸显了NIS-Seq的可靠性。结果显示,在高密度条件下,NIS-Seq的条形码映射率达到96.6%,显著高于传统的原位测序方法。同时,特异性与灵敏度的测试表明,NIS-Seq在不同固定方法下(如甲醇和PFA固定)都能保持高水平的信号特异性(接近100%)和低噪声背景。此外,通过对混合细胞群进行测试,研究人员发现NIS-Seq能够清晰地区分表达不同sgRNA的细胞,几乎没有误报或信号重叠现象。
更重要的是,NIS-Seq还能高效处理高复杂性基因文库。在多轮测序的实验中,它能够区分超过69,000种基因条形码,与下一代测序(NGS)结果的相关性高达0.863。这种能力使得研究人员在基因文库覆盖率和准确性上达到了前所未有的高度。
通过这些数据可以看出,NIS-Seq不仅在高密度筛选中具备优越性能,还极大地提升了基因扰动分析的准确性和稳定性。
从炎症通路到巨噬细胞:揭示关键基因的秘密
炎症反应是免疫系统应对病原体和组织损伤的核心机制,而解码炎症通路中基因的具体作用,一直是现代医学研究的重要课题。NIS-Seq技术的问世,为这一复杂生物过程提供了全新的解析工具。该研究中,研究人员利用NIS-Seq对炎症通路的关键基因进行了系统性筛选,揭示了多个基因在炎症调控中的核心作用,并在原代巨噬细胞的研究中取得了开创性突破。
在实验中,研究人员以HeLa细胞为模型,通过NIS-Seq技术解码了NF-κB信号通路这一经典炎症通路的基因作用机制。NF-κB是一组诱导性转录因子家族,其活化过程涉及复杂的信号传导网络。研究人员通过CRISPR文库对超过18,000个基因进行了全基因组水平的扰动,并使用NIS-Seq结合荧光标记的p65蛋白对细胞核的信号转导进行了精准量化。筛选结果确认了IL1R1、TNFRSF1A等经典受体基因,同时也揭示了多个新基因(如TRAF6、CHUK)的作用,进一步完善了NF-κB通路的基因网络图。
更值得一提的是,该研究首次成功将NIS-Seq应用于原代人巨噬细胞的基因筛选。这种细胞类型生物学意义重大,但由于其低转录活性、难以转染等特点,一直难以被传统筛选技术有效研究。研究人员通过优化CRISPR载体与NIS-Seq技术,成功完成了巨噬细胞中NLRP3炎症小体(inflammasome)的筛选实验。结果显示,NLRP3、IKBKB和IRAK1等基因对炎症小体的形成和功能至关重要,同时揭示了若干新基因(如HDAC5、RNF31)的调控作用,为理解炎症小体的分子机制提供了新视角。
这项研究的重要性在于,NIS-Seq不仅揭示了经典炎症通路中新的基因调控机制,还填补了原代巨噬细胞筛选的技术空白。通过这种创新性的基因筛选手段,研究人员不仅能够深入理解炎症通路的复杂网络,还为治疗炎症性疾病提供了潜在的新靶点。
首次尝试:人皮肤组织中的条形码识别
在复杂的活体组织中进行基因筛选一直是生物医学研究的前沿挑战之一。相比于细胞培养体系,组织中的细胞具有高度复杂的空间结构和多样的生理状态,这对传统筛选技术提出了严峻的挑战。NIS-Seq技术的出现,不仅突破了单细胞筛选的限制,更首次成功实现了在活体组织中对基因扰动条形码的精确识别,为组织水平的遗传研究开启了新篇章。
研究人员在人皮肤组织中进行了这一前沿实验。他们首先从健康捐献者的皮肤活检样本中分离出上皮细胞层,并用经过优化的CRISPR载体库对其进行转导。这些载体不仅携带了NIS-Seq兼容的条形码,还编码了炎症相关信号通路的基因扰动因子。随后,研究团队利用NIS-Seq技术在经过多轮荧光测序后,精准识别了每个细胞核内的条形码序列,成功将基因扰动信息映射到皮肤组织的细胞层。
在多轮条形码测序中,NIS-Seq信号显示了高度的特异性和可靠性,能够清晰地区分转导细胞和未转导细胞,且没有出现显著的信号重叠或背景噪声。此外,研究人员还通过组织变形补偿算法,成功解决了活体组织在固定和测序过程中可能产生的图像畸变问题。这些技术优化使得条形码的空间映射达到了前所未有的精准度。
这一突破意义深远。首次在人皮肤组织中实现条形码的精确识别,不仅展示了NIS-Seq在活体组织研究中的应用潜力,也为未来深入研究组织水平的基因功能和疾病机制奠定了基础。例如,通过这一技术,研究人员未来可在复杂的组织微环境中系统性地研究基因扰动对细胞行为的影响,甚至进一步探索疾病组织中的遗传调控网络。
光学筛选的未来
随着生物学研究逐步迈向大数据时代,如何将高通量数据转化为深刻的生物学洞察,成为科学界面临的核心挑战之一。NIS-Seq技术的问世,为这一挑战提供了前所未有的解决方案。它不仅能够在单细胞水平上生成海量的基因扰动数据,还能通过结合多维度的高通量分析,深入挖掘复杂的生物学问题,揭示潜藏在细胞和组织中的遗传机制。
NIS-Seq技术以其超高通量和分辨率,使研究人员能够在短时间内生成覆盖数十万细胞和数万基因条形码的数据集。例如,在对NF-κB信号通路的筛选中,NIS-Seq成功解析了超过18,000个基因的扰动效果,并通过精确的单细胞核信号量化,构建出一幅详细的基因功能网络图。在炎症小体研究中,NIS-Seq进一步揭示了许多未被充分研究的调控因子,为深入理解炎症反应的分子机制提供了关键线索。
此外,NIS-Seq的强大之处在于其能够同时捕获单细胞分辨率的表型和基因型数据,并通过统计分析和机器学习技术,从复杂数据中提取关键特征。例如,研究人员利用NIS-Seq生成的超过70万张单细胞图像,采用零知识嵌入(zero-knowledge embedding)算法,成功挖掘了传统筛选方法中被遗漏的关键基因(如RELA),展现了人工智能与光学筛选结合的强大潜力。
未来,NIS-Seq技术的应用范围远不止于实验室研究。其能够以高精度解析基因功能的能力,在临床研究中具有广阔前景。例如,NIS-Seq可用于精准筛选疾病相关基因,为癌症、免疫性疾病等提供潜在的治疗靶点。此外,该技术在组织水平的高效应用,预示着它将在组织工程、药物筛选和再生医学中扮演重要角色。
NIS-Seq通过其独特的光学筛选能力,正在为基因组学研究开辟新的路径。从单细胞到组织,从实验室到临床,NIS-Seq为研究人员提供了一个强大的平台。
下一步在哪里?
尽管NIS-Seq技术以其独特的核内信号生成和高通量筛选能力,推动了基因组学和生物医学研究的前沿,但它仍存在一些局限性需要进一步改进。
首先,NIS-Seq的信号检测效率仍有提升空间。研究中发现,大约40%至60%的细胞核能够被成功映射到基因库中的条形码。这一限制可能源于几个因素:例如,有些细胞可能未能成功转导完整的CRISPR载体,或者条形码在病毒包装和转导过程中发生了突变。此外,在高密度筛选中,尽管NIS-Seq在单细胞分辨率上的表现优异,但对于部分多重转导细胞或条形码重叠现象,仍可能出现信号噪声或识别错误。
其次,NIS-Seq的实验过程较为复杂,需要严格控制固定和测序步骤。例如,不同的固定方法(如甲醇或PFA)对信号特异性的影响尚需进一步优化。此外,尽管该技术已成功应用于组织研究,但在更复杂的三维结构和动态组织环境中的表现仍需验证,这对技术的稳定性和适配性提出了更高的要求。
针对这些挑战,未来的改进方向可能包括优化CRISPR载体设计以提高转导效率和条形码完整性,同时探索新型信号放大技术(如CoolMPS抗体标记),以进一步提升信号强度和降低噪声。此外,自动化实验流程和人工智能算法的引入,也将有助于提高数据处理效率和结果的准确性。
在潜在应用领域方面,NIS-Seq展现了广阔的前景。从基础研究到临床应用,该技术可用于解码更多复杂的遗传网络,如癌症中的多基因协同效应或神经退行性疾病的关键调控因子。在再生医学领域,NIS-Seq还可用于探索干细胞分化和组织修复的动态机制。
总的来说,NIS-Seq技术虽尚处于发展初期,但它的出现已经为基因组学和生物医学研究打开了新的窗口。随着技术的不断迭代,我们有理由相信,NIS-Seq将在未来释放更大的潜力,成为生命科学研究不可或缺的核心工具之一。
参考文献
Fandrey, C.I., Jentzsch, M., Konopka, P. et al. NIS-Seq enables cell-type-agnostic optical perturbation screening. Nat Biotechnol (2024). https://doi.org/10.1038/s41587-024-02516-5
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言
















前往app查看评论内容
0 0
前往app查看评论内容
0 0
前往app查看评论内容
2 0