Nature:细胞命运重塑的新篇章:染色质结构与重编程因子的协奏
23小时前 生物探索 生物探索 发表于陕西省
文章介绍细胞命运转换的意义与问题,重点讲 Nature 报道的研究,涉及转录因子结合、染色质结构,还阐述细胞重编程技术的应用前景。
引言
细胞命运的转换是现代生命科学和医学领域中最具革命性的话题之一。通过精准操控细胞命运,研究人员可以将一种成熟的细胞类型重编程为另一种完全不同的细胞类型,这种过程不仅揭示了生命系统的可塑性,更为再生医学、疾病建模和个性化治疗开辟了新的可能性。然而,重编程的核心机制依然存在许多未解之谜:转录因子(Transcription Factors, TFs)如何从庞大的基因组中快速而精准地找到目标结合位点?在高度压缩的染色质结构中,它们又是如何协同打开特定基因的表达开关以引导细胞命运的改变?
近年来,研究人员发现,一些特殊的转录因子,称为先锋因子(pioneer factors),能够直接结合于“关闭”的染色质区域,并重新塑造其开放状态,吸引其他因子的进入,从而激活基因表达。然而,仅凭对DNA序列基序(motif)的识别,尚不足以解释这些因子在基因组上的选择性结合行为。这种现象表明,染色质三维结构(3D chromatin organization)和核小体的拓扑结构(nucleosome fibre topology)可能扮演了重要角色。12月18日Nature的研究报道“Nucleosome fibre topology guides transcription factor binding to enhancers”,正是针对这一关键问题展开,通过解析不同转录因子组合在核小体上的结合规律,探讨了染色质拓扑结构如何指导因子的协同结合与细胞命运决定。
该研究采用了多种转录因子组合,包括经典的OSKM因子(OCT4、SOX2、KLF4、MYC)和用于滋养干细胞(trophoblast stem cells)诱导的GETM因子(GATA3、EOMES、TFAP2C、MYC),对小鼠胚胎成纤维细胞(MEFs)进行了重编程实验。通过结合转录组学、染色质免疫共沉淀测序(ChIP-seq)及微核酸酶消化测序(MNase-seq)等技术,研究团队深入剖析了不同因子组合如何以特定的模式绑定核小体,并揭示了这些模式如何影响基因的表达与细胞命运的塑造。这项工作不仅揭示了细胞重编程背后的分子密码,还提出了一种全新的模型:基于核小体拓扑结构的“引导搜索”(guided search)机制。
这一发现为我们理解细胞命运的基础规则提供了新的视角,同时也为未来的干细胞研究、基因治疗以及疾病建模提供了理论依据和技术方向。通过打开染色质的“密码”,我们正在逐步揭示生命塑造的秘密,并朝着操控细胞命运的目标迈进。

打开细胞命运之锁:细胞重编程的科学革命
生命的奥秘,往往蕴藏在细胞的命运之中。每个细胞似乎从诞生起就被赋予了固定的角色,例如心肌细胞负责跳动,神经细胞负责传递信号。然而,研究人员发现,这种“命运”并非不可逆转。细胞重编程技术的诞生,正是为了解锁这一自然规律,赋予细胞“再选择”的权利。通过引入特定的转录因子(Transcription Factors, TFs),原本分化成熟的体细胞可以被重新塑造成其他类型的细胞,甚至重回胚胎干细胞(Pluripotent Stem Cells, PSCs)的状态。这一技术的出现,不仅为再生医学打开了崭新的大门,也重新定义了人类对细胞命运的理解。
细胞重编程的历史,可以追溯到上世纪70年代。当时的研究人员首次通过核移植实验表明,细胞核中的遗传信息具有全能性(totipotency)。这一发现的真正突破发生在2006年,日本科学家山中伸弥及其团队通过引入四个关键转录因子——OCT4、SOX2、KLF4和MYC(统称OSKM),成功将小鼠体细胞重编程为诱导多能干细胞(iPSCs)。自此之后,细胞重编程领域迅速发展,不同的转录因子组合被开发出来,例如GETM因子(GATA3、EOMES、TFAP2C、MYC),能够将细胞重编程为诱导滋养干细胞(iTSCs)。这些突破性的发现不仅揭示了转录因子在重编程中的核心作用,也为理解细胞命运调控提供了宝贵的线索。
细胞重编程的意义,早已超越了基础科学的范畴。在再生医学中,它为治疗退行性疾病、创伤修复和器官移植提供了巨大的潜力;在疾病建模中,它为研究罕见疾病和个性化药物开发提供了新工具;甚至在生物制造领域,重编程技术还可能催生出功能化细胞产品。然而,重编程的过程远非简单的基因“开关”操作。
先锋因子的奥秘:如何穿越关闭的染色质?
在细胞命运的旅程中,染色质的开放状态决定了哪些基因可以被激活,而哪些基因将被沉默。然而,大多数转录因子只能在“开放”的染色质区域工作,对“关闭”的染色质束手无策。这时,一种特殊的转录因子——先锋因子(Pioneer Factors)闪亮登场,它们能够突破关闭染色质的“防御”,为其他因子的到来打开通道,如同破译密码的先行者。
先锋因子的独特之处在于它们可以直接结合于核小体表面,而核小体通常是DNA缠绕在组蛋白上的压缩结构,是关闭染色质的基本单元。例如,OSKM因子中的OCT4和SOX2就具有这种先锋能力。在研究中,研究人员发现这些因子能够识别核小体上隐藏的特定基序(motifs),并通过与DNA结合改变染色质结构,使其更加“松散”。这种染色质重塑作用不仅为后续因子的结合提供了空间,还激活了关键基因的表达,从而推动细胞命运的改变。
数据显示,OCT4和SOX2等先锋因子在染色质关闭区域(closed chromatin)中的结合位置具有特异性。例如,研究发现,OCT4的结合基序通常位于核小体的超螺旋位置(SHL)3.5到6.5之间,而SOX2则更倾向于结合核小体核心区域。这些结合行为显示了先锋因子如何精准识别隐藏的目标,并依靠特定的“密码”解锁染色质。
更令人惊叹的是,先锋因子并非孤军作战。在细胞重编程过程中,它们通常与其他转录因子协同作用。例如,在OSKM组合中,OCT4和SOX2先行绑定于关闭染色质,随后吸引KLF4和MYC等因子的加入,从而完成染色质的全面重塑和基因激活。这种协同作用不仅显著提高了重编程效率,也展示了染色质打开过程中复杂而有序的机制。
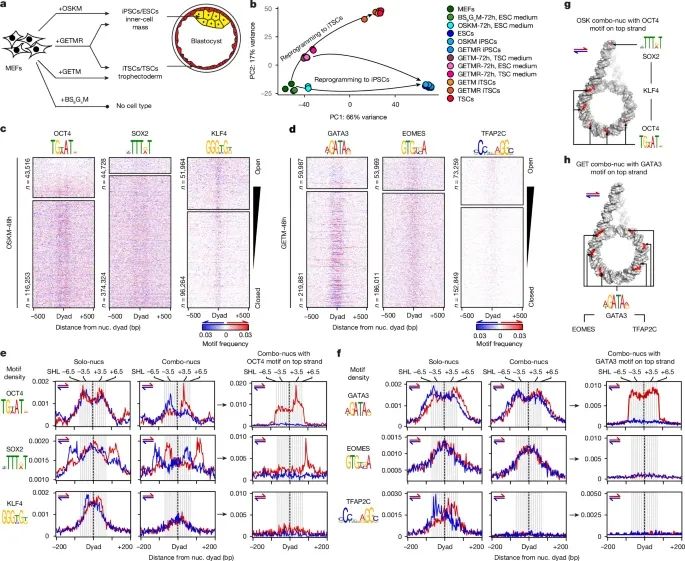
单核小体上的基序解读仅能解释转录因子(TF)的单独结合(Credit: Nature)
图a:胚泡模型与重编程因子的组合
展示了不同重编程因子组合(如OSKM、GETM和GETMR)诱导的细胞命运如何模拟胚泡发育中的不同细胞类型:OSKM组合可以将小鼠胚胎成纤维细胞(MEFs)重编程为诱导多能干细胞(iPSCs),类似胚泡中的内细胞团(inner cell mass)。GETM组合能够将MEFs重编程为诱导滋养干细胞(iTSCs),类似胚泡中的滋养层(trophectoderm)。GETMR因子组合的重编程路径因培养条件不同而分支,分别通向iPSCs或iTSCs。
图b:主成分分析(PCA)揭示重编程轨迹
使用RNA测序(RNA-seq)数据进行的PCA分析展示了OSKM和GETMR驱动的细胞重编程路径:OSKM因子直接将MEFs重编程为iPSCs。GETMR因子展示出双分支轨迹,根据培养条件决定细胞命运走向iPSCs或iTSCs。这种分支轨迹反映了不同因子组合对细胞命运的分化影响。
图c和d:OSKM和GETM因子的结合模式
图c展示了OSKM因子在开放和关闭染色质中的结合特点:OSKM因子的基序密度热图显示,OCT4、SOX2和KLF4在染色质开放(open chromatin)和关闭(closed chromatin)区域均可结合。基序密度呈现明显的方向性,说明OSKM因子对核小体的结合具有特定的空间偏好。图d与c类似,展示了GETM因子的结合特点:GATA3、EOMES和TFAP2C在染色质关闭区域中的结合基序分布更为显著,且基序排列方向也具有一定规律。
图e和f:单因子(solo-nucs)与多因子协同结合(combo-nucs)模式比较
图e(OSKM因子):单独结合:OCT4基序主要分布在核小体的超螺旋位置(SHL)3.5到6.5。协同结合:多个OSKM因子绑定同一个核小体时,其基序的方向性和分布更加集中,形成组合核小体(combo-nucleosome)。OSKM因子的“单独结合”(solo binding)与“协同结合”(combo binding)模式表现出显著差异,图中还显示了带有OCT4基序的核小体结构细节,强调了方向性在协同结合中的重要性。图f(GETM因子):GETM因子显示类似的模式,但GATA3和TFAP2C的方向性更为突出,而EOMES的结合模式较为分散。
图g和h:组合核小体的结构示意图
图g展示了OSKM组合核小体的结构:核小体上的OCT4基序(红色)位于顶层,与SOX2和KLF4基序形成协同结合的空间布局。图1h展示了GETM组合核小体的结构:核小体上的GATA3基序在顶层,其他因子如EOMES和TFAP2C的基序分布补充了协作结合的模式。
核小体上的“密码”:重编程因子如何找到目标?
在细胞重编程的过程中,转录因子需要在庞大的基因组中精准定位并绑定特定的DNA序列,这一过程看似偶然,却隐藏着一套精妙的“密码”。该研究揭示,这套密码不仅与DNA序列本身有关,还受到核小体的三维拓扑结构的严格控制。这些复杂的染色质结构如何成为转录因子结合的“路标”,正是研究人员试图破解的关键问题。
核小体是染色质的基本单位,由DNA缠绕在组蛋白八聚体上形成。然而,DNA在核小体中的排列并非随机,而是具有方向性和高度组织化的三维拓扑特征。研究发现,转录因子在结合DNA时,实际上是在解读核小体的空间分布模式。数据显示,OSKM因子中的OCT4、SOX2和KLF4在核小体表面的结合基序(motifs)有着严格的空间限制:OCT4的基序通常分布在超螺旋位置(SHL)3.5到6.5之间,而SOX2的结合更接近核小体中心。这种精确的分布不仅有助于因子找到目标,也反映了核小体的三维结构对结合位点的指导作用。
更重要的是,当这些因子协同工作时,它们的结合模式发生显著变化。例如,在OSKM组合中,多个转录因子可以同时绑定同一个核小体,形成所谓的“组合核小体”(combo-nucleosome)。研究发现,这些组合核小体上的基序排列遵循特定的“方向性规则”,例如OCT4和SOX2基序在核小体上的分布呈现相反方向,而KLF4则集中于核心区域。这些方向性的排列使转录因子的协同作用更为高效,并最终推动基因的激活。
这一切的背后,是染色质三维结构作为“密码”的作用。已进入提出了一种“引导搜索”(guided search)模型,认为转录因子能够通过解读核小体的拓扑特征,精准找到目标区域。这种机制大大缩小了它们在基因组中“搜索”的范围,使得重编程过程更加快速和高效。
核小体的三维结构不再是单纯的染色质包装单位,而是转录因子结合的导航系统。
OSKM与GETM:两条重编程之路的密码解析
在细胞重编程的领域,OSKM与GETM是两组具有代表性的转录因子组合,它们如同两把截然不同的钥匙,分别解锁了细胞命运的两条道路:多能性(pluripotency)和滋养性(trophoblast fate)。尽管这两组因子都能重新塑造小鼠胚胎成纤维细胞(MEFs)的命运,但它们的作用方式、结合规律和细胞路径却截然不同。
OSKM因子由OCT4、SOX2、KLF4和MYC组成,是细胞重编程领域的“明星组合”。它们的任务是将成纤维细胞转变为诱导多能干细胞(iPSCs),一种与胚胎干细胞(ESCs)功能类似的细胞类型。研究表明,OSKM因子倾向于结合关闭染色质(closed chromatin),通过先锋因子OCT4和SOX2打开基因组中“沉睡”的区域,为KLF4和MYC的进入铺平道路。在核小体层面,OSKM因子的结合表现出明确的方向性和协同性。OCT4、SOX2和KLF4在核小体上的结合位点分布有一定规律,能够形成“组合核小体”(combo-nucleosome),这大幅提高了其打开染色质的效率,从而激活多能性基因。
相比之下,GETM因子(GATA3、EOMES、TFAP2C、MYC)的任务是将MEFs重编程为诱导滋养干细胞(iTSCs),一种与滋养层干细胞(TSCs)相似的细胞类型。与OSKM不同,GETM的结合模式更加复杂且具有分层性。研究发现,GETM因子在结合染色质时倾向于聚集在高度互联的核小体区域。这些区域富含GATA3和EOMES的基序,并形成具有两种方向性分类的核小体“阵列”。有趣的是,GETM的协作模式与OSKM有所不同,其基序分布更加均匀,方向性不如OSKM明显,但其结合区域的核小体更紧密。
这两组因子的结合规律直接影响了重编程路径的分化轨迹。主成分分析(PCA)显示,OSKM组合驱动细胞直接向多能性方向发展,而GETM则表现出更复杂的分支轨迹,不同的培养条件可决定细胞命运是多能性还是滋养性。这种路径分化反映了因子本身的结合特点和染色质重塑能力的差异。
OSKM与GETM的研究不仅为我们揭示了重编程的多样性,还提供了一个关于细胞命运可塑性的全新视角。
基序与拓扑的协奏曲:染色质结构如何“引导搜索”?
在基因组中,转录因子如何从几十亿个碱基对中精准找到自己的结合位点?这不仅是细胞重编程中需要回答的核心问题,也是解码基因调控网络的关键。该研究提出了一种全新的“引导搜索”(guided search)机制,揭示了染色质结构如何为转录因子的结合提供导航,并大幅提高结合效率。这一发现将基序识别和染色质拓扑结构的作用完美结合,如同谱写了一曲生物分子合作的“协奏曲”。
在传统认知中,转录因子的结合主要依赖DNA序列中的特定基序(motifs)。然而,研究显示,单凭基序识别不足以解释转录因子的高效结合。该研究的数据进一步揭示,核小体的三维拓扑结构提供了额外的空间线索。通过微核酸酶消化测序(MNase-seq)和染色质免疫共沉淀测序(ChIP-seq),研究发现,OSKM因子在染色质上并非随机分布,而是受到核小体排列方向性的精确引导。例如,OCT4和SOX2的基序在核小体阵列中呈现出明显的方向性,且这些阵列通常位于染色质环(chromatin loops)的边界处,为转录因子的结合提供了最佳的立体位置。
更进一步,研究提出,这种“引导搜索”机制不仅依赖单个核小体的基序分布,还涉及更大范围的核小体阵列和染色质。实验显示,OSKM因子首先绑定于核小体阵列的“近端边界”,随后沿着阵列方向性分布,逐步扩展至基因增强子(enhancers)。这种拓扑特性犹如“路标”,为转录因子的协同结合指明了方向,从而避免了随机搜索所带来的时间和能量浪费。
在GETM因子中,这一机制表现出不同的特性。GETM组合的转录因子更倾向于结合在染色质环的交汇点,这些位置被称为“高度互联的路标元素”(highly connected signpost elements)。这些区域的核小体排列更加紧密,具有明显的边界基序(例如GATA3和TFAP2C的基序),从而进一步优化了转录因子的结合效率。
“引导搜索”机制重新定义了转录因子结合的规则。它通过基序的方向性排列和染色质拓扑结构,缩小了基因组中转录因子需要探索的空间范围。这种机制不仅揭示了染色质结构对基因表达调控的重要性,也为未来优化细胞重编程效率和基因治疗技术提供了新的策略。
重塑生命:从实验室走向应用的重编程技术
细胞重编程技术不仅揭示了细胞命运的可塑性,更为医学和生物技术的应用提供了无限可能。通过控制特定转录因子的组合与染色质结构,研究人员正在探索如何将这项技术从实验室的发现转化为解决临床问题的强大工具。这项技术的前景如同重新书写生命的语言,将影响从再生医学到疾病建模的方方面面。
首先,在再生医学中,细胞重编程带来了修复和再生受损组织的新方法。例如,通过OSKM因子将体细胞重编程为诱导多能干细胞(iPSCs),可以生成无限增殖的干细胞。这些细胞能够进一步分化为心肌、神经或肝细胞,为治疗退行性疾病(如帕金森病、阿尔茨海默病)和心肌梗死等病症带来了新的可能性。此外,GETM因子组合能够生成诱导滋养干细胞(iTSCs),为妊娠相关疾病和胎盘功能障碍的研究与治疗提供了独特的细胞来源。这些重编程技术消除了传统胚胎干细胞存在的伦理争议,同时为个性化再生治疗开辟了新路径。
其次,在基因治疗领域,重编程技术也展现出巨大的潜力。研究显示,转录因子与染色质的精确结合能够有效激活或沉默特定基因,从而为治疗单基因遗传病或复杂疾病(如癌症)提供可能。例如,通过理解OSKM和GETM因子在染色质中的“引导搜索”机制,可以开发新的基因编辑策略,使治疗靶点更加精准,副作用更少。
最后,细胞重编程在疾病建模中的应用令人瞩目。通过将患者体细胞重编程为特定类型的干细胞,研究人员可以在实验室中模拟疾病的发生与发展,从而加速药物筛选和治疗方案的优化。例如,利用iPSCs建立神经退行性疾病的模型,可以揭示疾病的分子机制,并测试多种潜在药物的效果。这种个性化疾病建模方法为精准医学奠定了基础。
该研究不仅为我们提供了理解转录因子与染色质相互作用的分子图谱,更为这些技术的临床转化奠定了理论基础。重编程技术正从实验室的试验走向实际应用,它赋予了细胞新的命运,也赋予了生命科学新的可能性。
参考文献
O’Dwyer, M.R., Azagury, M., Furlong, K. et al. Nucleosome fibre topology guides transcription factor binding to enhancers. Nature (2024). https://doi.org/10.1038/s41586-024-08333-9
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言














#转录因子# #细胞重编程#
4