最新Nature大子刊:大幅减少基因编辑副作用!
2024-11-10 BioMed科技 BioMed科技 发表于上海
本研究发现 Cas9 核酸酶易产生大基因组缺失,研究优化测序和算法,对比 Cas9、碱基编辑器和素数编辑器,揭示产生大缺失机制,为避免问题提供见解。
当实行基因组编辑时,Cas9核酸酶在DNA中会产生靶向双链断裂。后续的DNA修复途径可能导致较大的基因组缺失(大于100 bp),而这限制了基因组编辑的适用性。
在此,首尔大学Sangsu Bae等人发现Cas9介导的双链断裂在癌症细胞系、人类胚胎干细胞和人类原代T细胞中以不同频率诱导较大的缺失,并且大多数缺失是由两种修复途径产生的:末端切除和DNA-聚合酶-θ介导的末端连接。这些发现需要优化长程扩增子测序,开发用于同时分析大DNA缺失和小DNA改变的k-mer比对算法,以及使用CRISPR干扰筛选。尽管利用了产生单链断裂的突变Cas9连接酶,碱基编辑器和素数编辑器也产生了较大的缺失,但频率比Cas9低约20倍。相关工作以“Large DNA deletions occur during DNA repair at 20-fold lower frequency for base editors and prime editors than for Cas9 nucleases”为题发表在Nature Biomedical Engineering。

【文章要点】
尽管CRISPR核酸酶产生的大多数缺失都短于20个碱基对(bp),但有研究观察到CRISPR-Cas9诱导的小鼠胚胎中超过20%的突变是长度超过250 bp的意外缺失。此外,CRISPR核酸酶诱导的DSB可以诱导染色体重排,包括染色体耗竭和易位。CRISPR-Cas9系统的另一种变体是使用碱基编辑器(BE)和素数编辑器(PE),它们使用部分失活的Cas9(nCas9),产生单链缺口。然而,这些基于nCas9的基因组编辑系统是否也会产生大的缺失尚不清楚。识别在用Cas9或nCas9进行基因组编辑后产生大缺失的修复途径,不仅可以提供对这一过程生物学的关键见解,还可以制定缓解缺失事件的策略。然而,使用短程高通量测序方法确定>100 bp的缺失事件频率具有挑战性。目前,这些事件是通过使用短读测序仪(如Illumina平台)的长程扩增子测序方法或使用太平洋生物科学公司(PacBio)或牛津纳米孔技术公司(ONT)平台的长读测序方法来检测的。而在这项研究中,作者改进了一种长程扩增子测序方法,并开发了一种新程序,使用名为Expanded Cas-Analyzer27(ExCas Analyzer)的专用k-mer比对算法同时分析CRISPR系统产生的大缺失和小突变,最终可准确地确定了CRISPR诱导的大缺失(图1)。
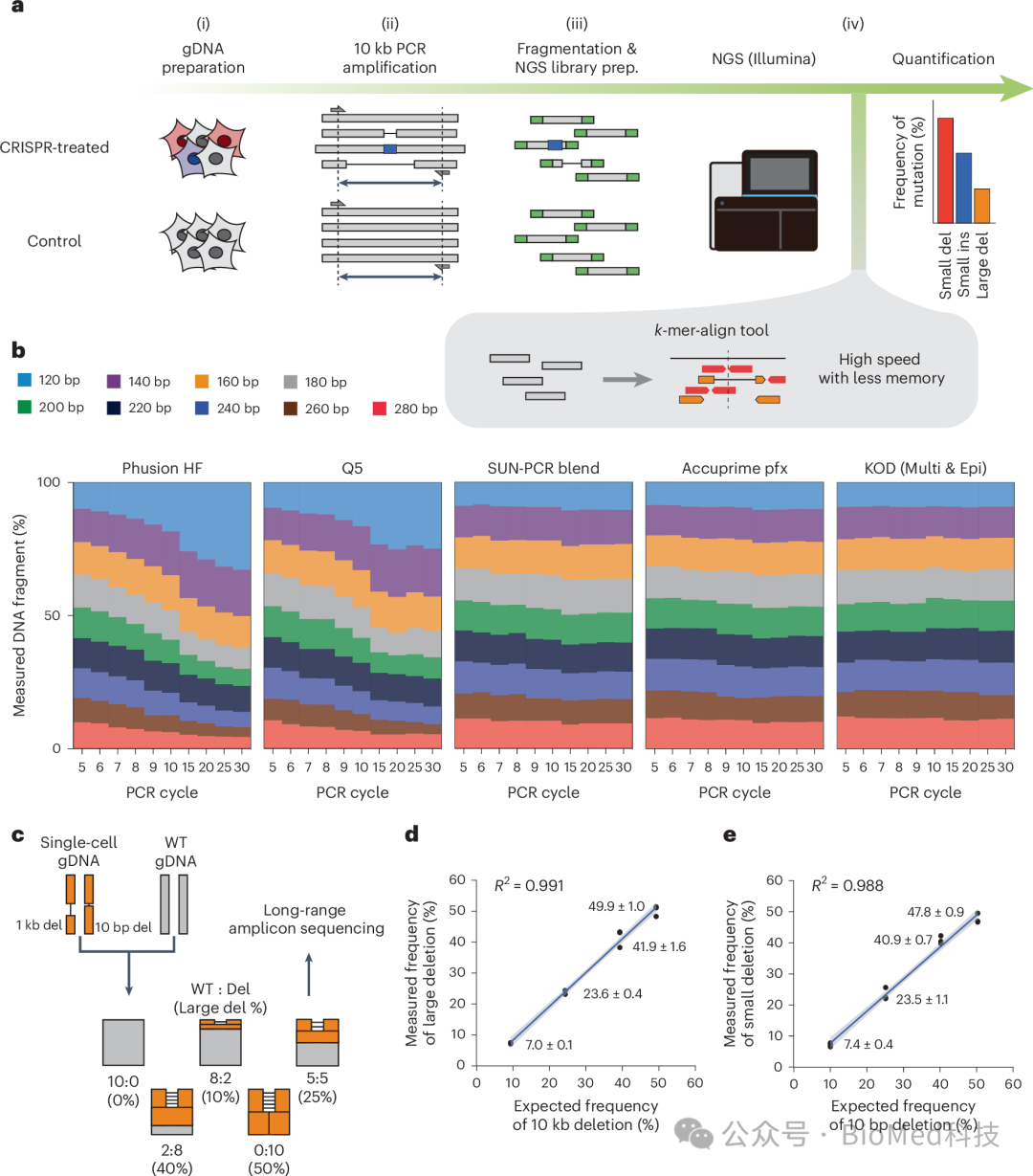
图1 远程深度测序方案
研究观察到,在人类细胞系中,包括癌样细胞(HeLa、HEK293T、U2OS、K562)、成纤维细胞、原代T细胞和人类胚胎干细胞(H9),大的缺失大多伴随着小的indel。然后,作者通过使用ONT纳米孔测序的CRISPR干扰(CRISPRi)筛选实验确定了与大缺失相关的修复途径,确定TMEJ是产生大缺失的主要途径。研究发现,BE通过碱基切除修复(BER)途径产生大的缺失,特别是在存在无嘌呤/无嘧啶位点(AP位点)的情况下,PE在存在额外的切割导向RNA(ngRNAs)的情况下尤易产生大的删除。这些研究结果揭示了使用基因组编辑工具的机制挑战,并为在研究和治疗应用中避免这些问题提供了见解(图2)。
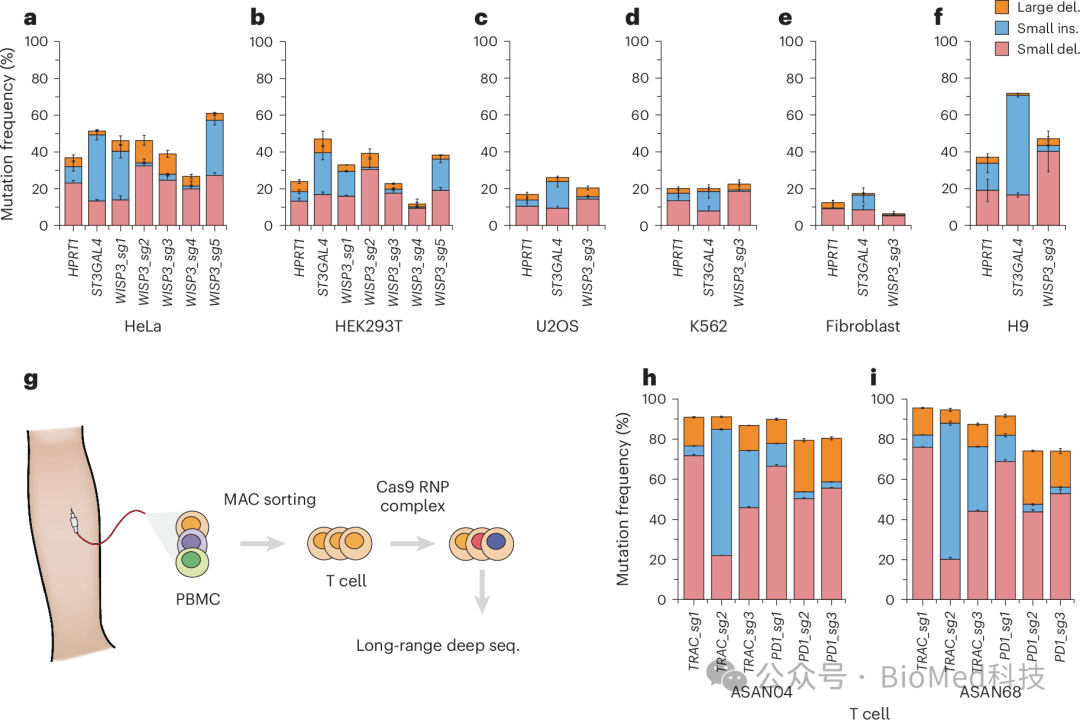
图2 不同细胞系的突变频率
原文链接:
https://www.nature.com/articles/s41551-024-01277-5
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言

















#基因组编辑# #碱基编辑器#
10