Nature Methods:基因变异与蛋白质功能的动态链接:G2P平台推动临床应用与药物开发的精准化
2024-09-20 生物探索 生物探索 发表于上海
G2P平台通过精确的基因-蛋白质映射工具,为结构生物学和基因组学的结合提供了极大的便利。
引言
近年来,随着人工智能(AI)技术的迅猛发展,结构生物学领域取得了显著进展。与此同时,高通量测序和功能基因组学的发展,使得我们能够生成前所未有数量的遗传变异。然而,如何有效地将这些基因变异数据与蛋白质序列和结构相联系,仍然是生物医学研究中的一大挑战。基因组变异的功能影响,尤其是与疾病相关的变异,往往依赖于它们在蛋白质结构中的具体位置。为此,研究者开发了“基因组到蛋白质”平台(Genomics 2 Proteins portal,G2P,https://g2p.broadinstitute.org/),这是一个用于将基因筛选输出与蛋白质序列和结构相连接的资源工具。该平台目前涵盖了20,076,998个遗传变异、42,413种蛋白质序列和77,923种蛋白质结构。(9月18日Nature Methods “Genomics 2 Proteins portal: a resource and discovery tool for linking genetic screening outputs to protein sequences and structures”)
G2P平台允许用户交互式上传蛋白质的残基注释(包括变异和打分),并且可以与公共数据库中的数据进行结合,如gnomAD(Genome Aggregation Database)、ClinVar和HGMD(Human Gene Mutation Database)。这一平台不仅能帮助研究者深入理解变异如何影响蛋白质的结构与功能,还为设计治疗方法提供了关键的线索。例如,研究者可以通过该平台假设特定蛋白质的结构与功能关系,从而为自然或合成变异的分子表型提供见解。
通过G2P平台,可以方便地检索并可视化基因-蛋白质的映射关系,并且能够将数据下载用于后续分析。这一工具为基因组学与蛋白质结构之间的桥梁提供了极大的便利,为探索与疾病相关的遗传变异的结构功能机制铺平了道路。

近年来,人工智能(AI)技术的进步极大地推动了结构生物学的发展,尤其是AlphaFold等AI驱动的蛋白质结构预测工具,为研究者提供了大量高精度的蛋白质结构模型。此外,高通量测序(High-Throughput Sequencing)技术的普及,使得我们能够更好地理解基因变异及其与疾病的关系。然而,尽管基因组学(Genomics)和功能基因组学(Functional Genomics)积累了大量的遗传变异数据,将这些变异数据与蛋白质序列和结构的精确对应仍然面临巨大挑战。
G2P平台( https://g2p.broadinstitute.org/ )集成了多种数据源,并通过用户友好的界面提供了多样化的变异分析工具。平台主要包含两大模块:“基因/蛋白质查询”(Gene/Protein Lookup)模块和“交互映射”(Interactive Mapping)模块。可以通过基因或蛋白质名称查询与该蛋白质相关的变异数据、序列信息及结构数据。此外,还可以上传自己的变异注释或功能评分数据,将其与目标蛋白质的结构相映射,从而为变异的结构-功能关系研究提供支持 。
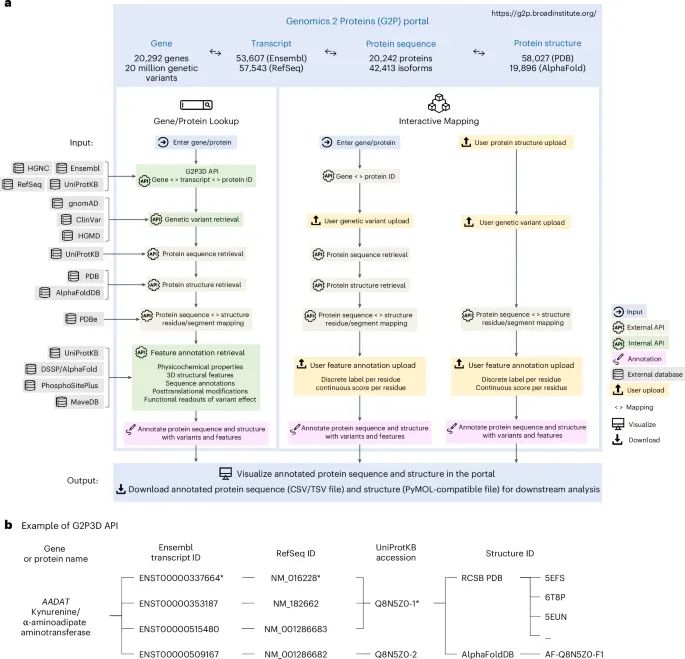
G2P平台的数据整合流程、功能模块和其动态交互式数据分析的核心框架(Credit: Nature Methods)
模块划分:G2P平台由两个主要模块组成:“基因/蛋白质查询”(Gene/Protein Lookup)和“交互映射”(Interactive Mapping)。这两个模块通过内部API (G2P3D API) 实现了从基因到蛋白质的动态映射。
数据整合:平台整合了多个数据库的数据资源,如gnomAD、ClinVar和HGMD,用于对基因变异的映射。同时,平台动态检索了UniProtKB、PDB和AlphaFoldDB中的蛋白质序列和结构信息,将变异注释到蛋白质序列和结构上。
可视化功能:用户可以通过平台上传自己的蛋白质残基注释(如变异和功能得分),并将这些数据与现有的数据库信息进行交互式映射。此外,所有注释的数据都可以在平台上进行可视化和下载,用于后续分析。
API功能:G2P3D API允许通过基因、转录本、蛋白质序列和结构的标识符,实现从基因变异到蛋白质结构的无缝映射,从而帮助研究者分析变异对蛋白质功能的影响。
平台基于React.js前端和Google Cloud后端架构,支持用户与广泛的数据资源交互。通过集成gnomAD(Genome Aggregation Database)、ClinVar和HGMD(Human Gene Mutation Database)等数据库,G2P平台涵盖了超过2000万个基因变异和超过77,000种蛋白质结构 。此外,可以上传与蛋白质残基相关的变异或注释信息,并将其映射到蛋白质结构上,进一步帮助分析变异对蛋白质功能的影响 。
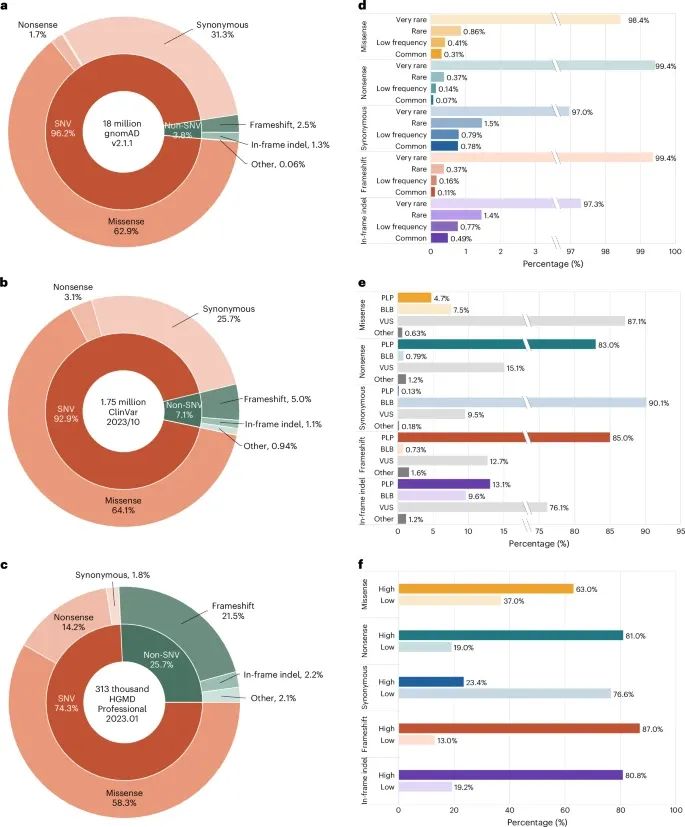
通过对变异类型、频率和临床意义的统计,展示了G2P平台如何将不同来源的变异数据汇总(Credit: Nature Methods)
变异类型分布:
gnomAD数据库:大多数蛋白质编码变异是单核苷酸变异(SNV),占96.2%,其中错义突变(missense)占比最高,为62.9%。此外,无义突变(nonsense)、同义突变(synonymous)、移码突变(frameshift)和其他类型的变异占比较低。
ClinVar数据库:主要变异类型也是SNV,错义突变占64.1%。无义突变、同义突变和移码突变的比例分别为3.1%、25.7%和5%。
HGMD数据库:同样以SNV为主,错义突变占58.3%,无义突变和移码突变分别占14.2%和21.5%。
变异频率分布:
gnomAD数据库:大多数变异是非常罕见的,约97%的变异频率低于0.1%。
ClinVar数据库:根据临床意义进行分类,大多数同义突变(90.1%)被归类为良性或可能良性(B/LB),而无义突变(83%)多为致病或可能致病(PLP)。
HGMD数据库:高置信度的疾病突变主要集中在移码突变和无义突变,分别占87%和81%。
变异的临床意义:
ClinVar数据库中的变异按其临床意义进行分类,错义突变的变异有很高比例属于不确定意义(VUS)或存在解释冲突的类别。HGMD中的变异按置信度划分,高置信度的疾病突变多为无义和移码突变,而同义突变的置信度通常较低。
G2P平台在多组数据的基础上进行了广泛的数据整合和分析。例如,通过整合gnomAD v2.1.1版本中的18,014,632个蛋白编码变异、ClinVar中的1,749,628个变异以及HGMD中的312,738个与疾病相关的突变,平台帮助用户深入分析变异在蛋白质序列和结构上的分布和功能影响 。其中,变异的分类包括错义突变(missense)、无义突变(nonsense)、同义突变(synonymous)、移码突变(frameshift)等,这些变异被映射到相应的蛋白质序列和结构上 。
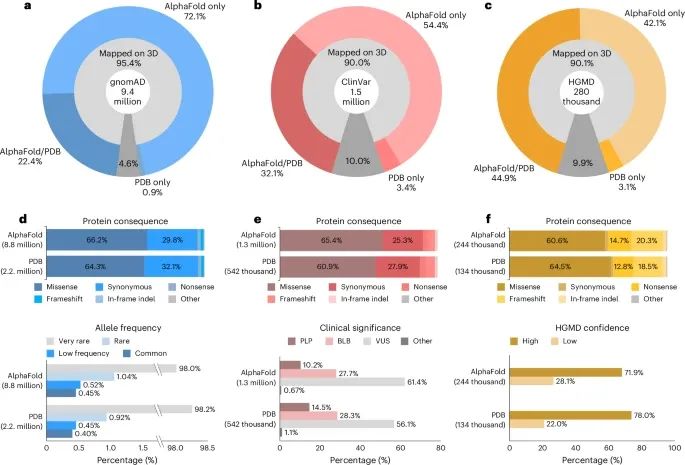
通过对变异映射到PDB和AlphaFold三维结构的详细统计,展示了G2P平台在变异功能分析中的应用,为研究者提供了精确的蛋白质结构功能分析工具,尤其是在分析遗传变异如何影响蛋白质结构和功能方面具有重要意义(Credit: Nature Methods)
变异映射到三维结构的比例:
gnomAD数据库:大约90%的gnomAD变异成功映射到PDB或AlphaFold结构上。其中54.4%的变异只映射到AlphaFold结构,32.1%映射到PDB和AlphaFold结构,3.4%仅映射到PDB结构。
ClinVar数据库:90.1%的ClinVar变异映射到三维结构,其中42.1%映射到AlphaFold结构,44.9%同时映射到PDB和AlphaFold结构,3.1%仅映射到PDB结构。
HGMD数据库:95.4%的HGMD变异映射到了三维结构,72.1%映射到AlphaFold结构,22.4%映射到PDB和AlphaFold结构,0.9%仅映射到PDB结构。
映射到PDB和AlphaFold结构上的变异分布:
gnomAD变异:无论是PDB还是AlphaFold结构,变异的分布差异不大。变异的蛋白质功能后果(如错义突变、同义突变等)在两种结构中的分布相似。
ClinVar变异:临床意义为致病/可能致病(PLP)的变异更多地映射在PDB结构上,比例为14.5%,而AlphaFold结构上仅为10.2%。同样,高置信度的致病突变更多出现在PDB结构中。
HGMD变异:高置信度的疾病相关突变有78%映射到PDB结构,而AlphaFold结构上仅有72%。
不同变异类型在三维结构中的分布:
无论是gnomAD、ClinVar还是HGMD,错义突变(missense mutations)是最主要的变异类型,其他类型如无义突变和移码突变则相对较少。
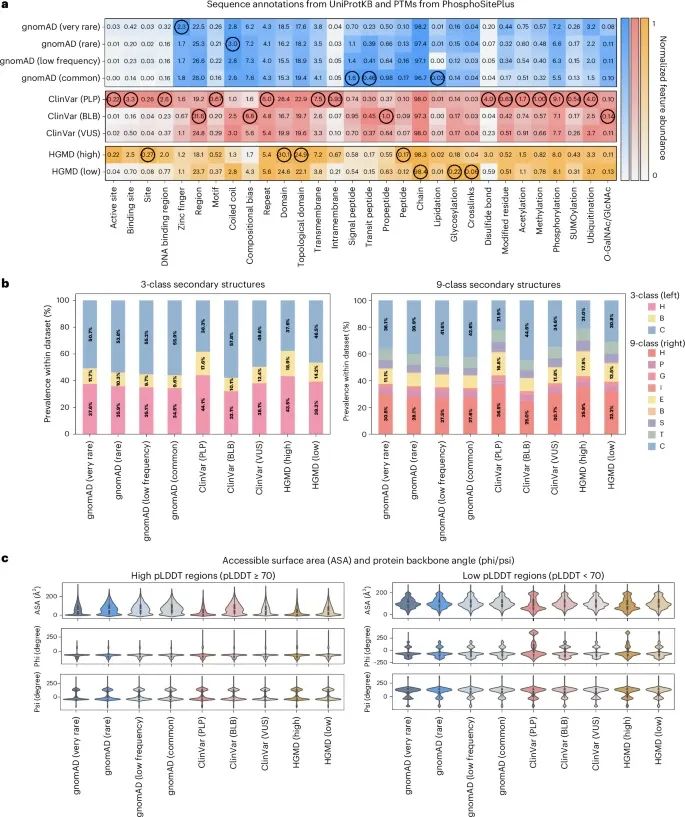
通过对蛋白质特征的丰度分析,展示了不同数据库中的错义变异如何分布于蛋白质的关键功能区域(Credit: Nature Methods)
蛋白质特征的丰度:该图比较了不同数据集中,基于UniProt的蛋白质序列注释和翻译后修饰(Post-Translational Modifications, PTMs)的丰度。这些蛋白质特征包括活性位点(active site)、结合位点(binding site)、DNA结合区域(DNA-binding region)、结构域(domain)等。数据显示,ClinVar数据库中具有致病/可能致病(P/LP)错义变异的数据集中,活性位点的丰度最高,表示这些变异更有可能出现在功能重要的蛋白质区域。相比之下,gnomAD中的常见变异(common variants)则在这些重要功能区域的丰度较低。
二级结构类型的分布:图中将二级结构分为三类:螺旋(helix)、链(strand)和环(loop)。ClinVar和HGMD数据库中的致病变异主要集中在结构较为稳定的螺旋和链区域,约56%的ClinVar P/LP变异和HGMD高置信度疾病突变出现在这些区域。而gnomAD中的常见变异更多出现在不太稳定的环区域。
三维结构特征的分布:图中展示了不同数据集中蛋白质残基的可接触表面积(accessible surface area, ASA)和骨架二面角(phi/psi angles)的分布情况。使用AlphaFold预测的高置信度区域(pLDDT ≥ 70)内的变异比低置信度区域(pLDDT < 70)的变异具有更集中的ASA和二面角分布,说明高置信度区域的结构更加稳定,这些区域的变异可能对蛋白质功能影响更大。
错义变异组中的特征比较:不同数据库中错义变异组的蛋白质特征存在显著差异。gnomAD数据库中的常见变异往往出现在不太重要的功能区域,而ClinVar PLP和HGMD高置信度变异则集中在更具功能意义的区域,如活性位点和结构域。这些差异有助于研究者识别可能对蛋白质功能有显著影响的突变位点。
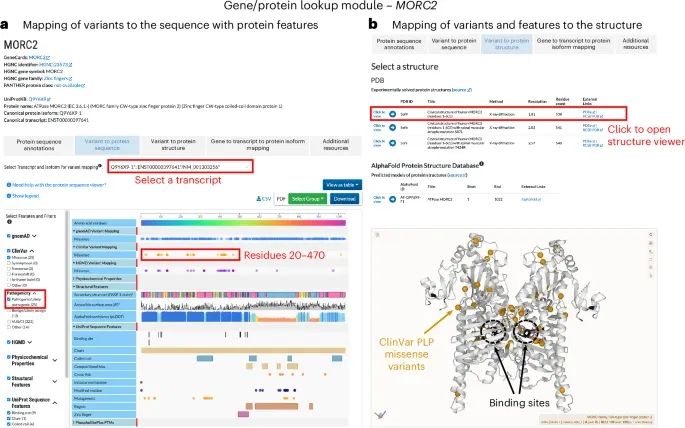
使用“基因/蛋白质查询模块”(Gene/Protein Lookup module)对MORC2蛋白已报道的变异和蛋白质特征的实际应用案例(Credit: Nature Methods)
蛋白质序列中的变异映射:可以在“基因/蛋白质查询模块”中选择MORC2基因,并查看其相关的蛋白质序列、变异和功能特征。该图展示了MORC2蛋白N端区域(残基20-470)中的多个ClinVar数据库中的致病/可能致病(PLP)错义变异。这些变异在蛋白质序列上以黄色标记展示,帮助识别这些重要的致病性变异。
蛋白质功能特征的映射:在蛋白质序列视图中,可以选择不同的功能特征进行展示,例如MORC2蛋白的N端区域含有多个结合位点(binding site)和催化位点(如ATP-和锌离子结合位点)。这些功能特征与ClinVar的致病性变异相重叠,提示这些变异可能通过影响结合位点或催化活性来干扰MORC2的正常功能。
三维结构中的变异映射:在“变异到蛋白质结构”选项中,可以将变异映射到MORC2的三维结构(PDB ID: 5OF9)。图中展示了ClinVar P/LP变异(黄色标记)如何在蛋白质的同二聚体结构上分布,并且这些变异靠近蛋白质的二聚界面和结合位点(以黑色标记)。这些信息帮助研究者理解这些变异可能通过改变蛋白质的二聚化或功能位点的结合能力来导致疾病。
以DNA甲基转移酶3A(DNMT3A)基因为例,研究者通过G2P平台将34个错义变异与蛋白质结构结合,揭示了这些变异对甲基转移酶和附加结构域(ADD domain)的影响。在此基础上,研究者能够通过交互式映射工具更直观地查看这些变异的空间分布,并将其与已知的致病性变异进行对比分析 。结果显示,34个基因编辑后的变异位点中,有31个被预测为致病变异,这为深入理解这些变异的分子机制提供了重要依据 。
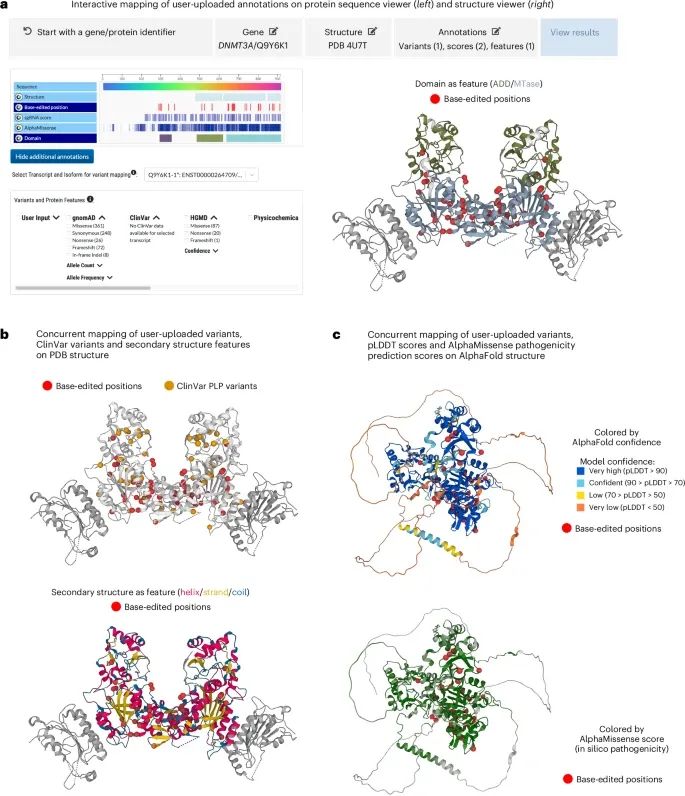
使用G2P平台的“交互映射模块”(Interactive Mapping module)对DNMT3A基因的碱基编辑(base-editing)结果的实际应用(Credit: Nature Methods)
变异注释的上传和映射:在交互映射模块中,可以通过输入基因/蛋白质标识符开始操作。在这个案例中,研究者选择了DNMT3A基因,并选择了其三维结构(PDB ID: 4U7T)。通过上传自己的注释数据,包括变异位点、连续数据(如得分)和离散特征(如功能域),并将其映射到蛋白质序列和结构上。图a展示了上传的34个错义变异(以红色小球表示)和两个结构域(Domain)的注释映射到DNMT3A蛋白的三维结构中。该模块支持同时查看多个注释,并根据需要选择不同的特征进行展示。
结合公开资源的变异分析:图b展示了上传的变异数据与G2P平台中提供的公开资源结合的结果。在这个例子中,通过同时查看基于ClinVar数据库的致病/可能致病(P/LP)变异(橙色小球)与上传的碱基编辑变异(红色小球)如何分布在DNMT3A的三维结构上。还可以选择二级结构特征(如螺旋、链、环)进行展示,以更好地理解变异在蛋白质中的空间分布。
变异与功能特征的共映射:图c展示了上传的变异如何与预测得分(如AlphaFold的pLDDT置信度和AlphaMissense的致病性预测得分)结合起来进行分析。在这一案例中,所有34个碱基编辑变异位点都映射在高置信度(pLDDT > 70)的蛋白质结构区域内,并且31个变异被AlphaMissense预测为致病变异(绿色表示较高的致病性得分)。这种分析可以帮助识别出潜在功能重要的变异位点。
G2P平台的开发为基因变异与蛋白质结构之间的关系研究提供了强有力的工具。平台不仅集成了多种公开数据库的数据,还允许用户上传自己的数据进行个性化分析,帮助研究者探索自然或合成变异的分子表型和功能机制。随着更多蛋白质结构数据和遗传变异数据的加入,G2P平台将在药物开发、靶点选择及精准医学领域发挥越来越重要的作用 。
总体而言,G2P平台通过精确的基因-蛋白质映射工具,为结构生物学和基因组学的结合提供了极大的便利。这一平台的应用将进一步推动遗传变异的功能研究,为疾病相关的机制研究提供新的思路。未来,随着蛋白质结构数据的进一步丰富,G2P平台在推动蛋白质功能研究中的作用将更加突出。
参考文献
Kwon S, Safer J, Nguyen DT, Hoksza D, May P, Arbesfeld JA, Rubin AF, Campbell AJ, Burgin A, Iqbal S. Genomics 2 Proteins portal: a resource and discovery tool for linking genetic screening outputs to protein sequences and structures. Nat Methods. 2024 Sep 18. doi: 10.1038/s41592-024-02409-0. Epub ahead of print. PMID: 39294369.
https://www.nature.com/articles/s41592-024-02409-0
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言


















#基因变异# #G2P平台# #蛋白质功能#
47