【论著】| 基于机器学习构建乳腺癌骨转移预测模型
10小时前 中国癌症杂志 中国癌症杂志 发表于陕西省
本研究选择9种机器学习算法探索建立有效的预测模型,并对模型评估对比,寻找更适合构建乳腺癌骨转移预测模型的方法。
[摘要] 背景与目的:乳腺癌是全球重大公共卫生问题,骨是乳腺癌远处转移最常见的部位,约占所有转移病例的70%。乳腺癌骨转移可引起一系列并发症,包括剧烈疼痛、病理性骨折、高钙血症、脊髓压迫等,给患者身体活动带来极大不便,影响生活质量。转移性复发是乳腺癌患者死亡的主要原因。因此迫切需要构建乳腺癌骨转移预测模型,以识别具有高骨转移风险的患者。本研究旨在开发基于机器学习的预测模型来预测乳腺癌发生骨转移的概率。方法:从监测、流行病学和最终结果(The Surveillance, Epidemiology, and End Results,SEER)数据库中提取2010年—2015年诊断的乳腺癌患者数据,并通过最小绝对收敛和选择算子(least absolute shrinkage and selection operator,LASSO)回归、单因素和多因素logistic回归分析对变量进行筛选,纳入具有统计学意义的风险因素构建预测模型。本研究使用决策树、弹性网络、 K最近邻、轻量级梯度提升机、logistic回归、神经网络、随机森林、支持向量机和极限梯度提升等9种机器学习算法,通过随机搜索和5倍交叉验证调整模型超参数,构建乳腺癌骨转移预测模型。利用受试者工作特征曲线(receiver operating characteristic,ROC)的曲线下面积(area under curve,AUC)、校准曲线和决策曲线对模型进行评价,得到最优模型,并基于最优模型分析变量的重要性。最后,应用最优模型建立预测乳腺癌骨转移风险的网络计算器。本队列研究严格遵循《加强流行病学中观察性研究报告质量》(Strengthening the Reporting of Observational Studies in Epidemiology,STROBE)指南中的各项条目。结果:本研究纳入10 106例乳腺癌患者,训练集7 073例患者,验证集3 033例患者,在这两个队列中,分别有4 494例(63.5%)和1 927例(63.5%)患者发生骨转移。种族、病理学分级、雌激素受体(estrogen receptor,ER)状态、孕激素受体(progesterone receptor,PR)状态、人表皮生长因子受体2(human epidermal growth factor receptor 2,HER2)状态、N分期、肺转移、放疗、化疗、手术是骨转移的独立预测因素。使用训练集和验证集对模型进行验证,综合ROC曲线的AUC、校准曲线和决策曲线等评价指标发现极限梯度提升算法优于其他机器学习算法。最后,本研究利用极限梯度提升算法构建预测乳腺癌骨转移的网络计算器,链接为https://bcbm.shinyapps.io/DynNomapp/。结论:本研究开发基于机器学习的预测模型,用于预测乳腺癌患者发生骨转移的概率,希望有助于临床医师作出更合理的治疗决策。
[关键词] 乳腺癌;骨转移;预测模型;机器学习;网络计算器
[Abstract] Background and purpose: Breast cancer is a major global public health problem. Bone is the most common site of distant metastasis of breast cancer, accounting for about 70% of all metastatic cases. Bone metastasis of breast cancer can cause a series of complications, including severe pain, pathological fracture, hypercalcemia, spinal cord compression, etc., which bring great inconvenience to patients' physical activities and affect their quality of life. Metastatic recurrence is the leading cause of death in breast cancer patients. Therefore, there is an urgent need to build a diagnostic model of bone metastasis in breast cancer to identify patients with a high risk of bone metastasis. The aim of this study was to develop a predictive model based on machine learning to predict the probability of breast cancer developing bone metastasis. Methods: Data of breast cancer patients diagnosed between 2010 and 2015 were extracted from The Surveillance, Epidemiology, and End Results (SEER) database. The variables were screened by least absolute shrinkage and selection operator (LASSO) regression, univariate and multivariate logistic regression analysis, and statistically significant risk factors were included to build a prediction model. In this study, nine machine learning algorithms, including decision tree, elastic network, K-nearest neighbor, lightweight gradient elevator, logistic regression, neural network, random forest, support vector machine and limit gradient lifting, were used to adjust the model hyperparameters through random search and 5x cross-validation to build a breast cancer bone metastasis prediction model. The area under the receiver operating characteristic (ROC) curve, calibration curve and decision curve were used to evaluate the model, the optimal model was obtained, and the importance of variables was analyzed based on the optimal model. Finally, a network calculator for predicting the risk of bone metastasis of breast cancer was established using the optimal model. Results: The study included 10 106 patients with breast cancer, 7 073 patients in the training set, and 3 033 patients in the validation set. We found that 4 494 (63.5%) patients in the training set and 1 927 (63.5%) patients in the validation set developed bone metastases, respectively. Race, pathologic grade, estrogen receptor (ER) status, progesterone receptor (PR) status, human epidermal growth factor receptor 2 (HER2) status, N stage, lung metastasis, radiotherapy, chemotherapy and surgery were independent predictors of bone metastasis. The training set and verification set were used to verify the model, and the limit gradient lifting algorithm was superior to other machine learning algorithms by integrating the evaluation indexes such as the area under the ROC curve, calibration curve and decision curve. Finally, we used limit gradient algorithm to build network calculator for prediction of breast cancer bone metastases (https://bcbm.shinyapps.io/DynNomapp/). Conclusion: This study developed a predictive model based on machine learning to predict the probability of bone metastases in breast cancer patients, hoping to help clinicians make more rational treatment decisions.
[Key Words] Breast cancer; Bone metastasis; Prediction model; Machine learning; Network calculator
2020年,女性乳腺癌在全世界估计有230万新病例,已超过肺癌成为最常见的诊断癌症[1]。在乳腺癌导致的死亡中,90%以上与转移有关[2]。骨、肺、肝、脑是乳腺癌的主要转移部位[3]。其中,骨转移最常见,发生率高达60%~70%[4]。最有效的癌症防治方法是预防和早期发现癌症[5]。因此,迫切需要识别具有高骨转移风险的患者,并寻找在癌症早期发生骨转移的预测因素。
机器学习有能力利用不同来源的数据作出准确预测,应用机器学习技术在医学领域对疾病诊断进行预测会给疾病防治带来益处[6]。近年来,机器学习已经运用到临床疾病诊断的各个领域,有研究[7]采用机器学习算法对皮肤癌进行分类,还有研究[8]使用机器学习对定期收集的电子健康纪录数据进行分析构建预测糖尿病前期至2型糖尿病的发展模型。
越来越多的电子医疗记录数据含有丰富的患者综合信息,如检查和诊断信息,加之机器学习的发展,为高效能预测模型的开发提供了新机会[9]。美国国家癌症研究所的监测、流行病学和最终结果(The Surveillance Epidemiology and End Results,SEER)数据库是一个开放的公共数据库,它来自18个人口注册网站,覆盖大约28%的美国人口,该数据库可以提供癌症患者的临床信息,例如性别、年龄、肿瘤原发部位、肿瘤分级、治疗措施等临床数据[10]。
本研究选择9种机器学习算法探索建立有效的预测模型,并对模型评估对比,寻找更适合构建乳腺癌骨转移预测模型的方法。我们还开发了基于最优机器学习模型的网络计算器,方便临床应用,以期实现对乳腺癌患者发生骨转移的早期预测,旨在为无创临床指标预测乳腺癌骨转移提供理论参考和临床指导。
1 资料和方法
1.1 数据来源
使用SEER*Stat 8.4.0.1软件,从SEER数据库2022年4月发布的“Incidence-SEER Research Plus Data, 17 Registries, Nov 2021 Sub(2000—2019)”中收集乳腺癌患者相关资料,末次随访时间为2021年11月。在开展这项研究之前,我们向SEER计划提交了数据使用协议,进而获得访问该数据库的权限。本研究符合《赫尔辛基宣言》要求,资料来源于开放公共数据库,免于伦理审查,不需要患者签署知情同意书。
1.2 纳入、排除标准
纳入标准:① 诊断年份为2010年—2015年;② 肿瘤原发部位为乳腺;③ M1期(远处转移)。排除标准:① 患者相关信息缺失或未知;② 总生存期小于1个月;③ 病理学检查结果由尸检或死亡证明获得;④ 随访信息不完整;⑤ 合并其他恶性肿瘤。
1.3 变量选择
纳入的变量包括年龄、种族、性別、婚姻状况、侧别、病理学分级、病理学类型、雌激素受体(estrogen receptor,ER)状态、孕激素受体(progesterone receptor,PR)状态、人表皮生长因子受体2(human epidermal growth factor receptor2,HER2)状态、分子亚型[ER和PR状态合并为激素受体(hormone receptor,HR)状态]、T分期、N分期、转移部位(骨、肝、肺和脑)、放疗情况、化疗情况、手术情况。
1.4 统计学处理
分类变量用频数(百分比)表示,分类变量比较采用χ2检验。使用R软件中随机抽样函数按7∶3比例分成训练集和验证集,采用“glmnet”包进行最小绝对收敛和选择算子(least absolute shrinkage and selection operator,LASSO)回归分析筛选变量,并对变量进行单因素和多因素logistic回归分析,得到独立风险因素;以此为基础在训练集中,采用“tidymodels”包使用决策树(decision tree,DT)、弹性网络(elastic net,EN)、K最近邻(K-nearest neighbor,KNN)、轻量级梯度提升机(light gradient boosting machine,LightGBM)、逻辑回归(light gradient boosting machine,LR)、神经网络(neural network,NN)、随机森林(random forest,RF)、支持向量机(support vector machine,SVM)和极限梯度提升(extreme gradient boosting,Xgboost)等9种机器学习算法,通过随机搜索和5倍交叉验证调整模型超参数,构建乳腺癌骨转移预测模型。本研究采用受试者工作特征(receiver operating characteristic,ROC)曲线的曲线下面积(area under curve,AUC)、校准曲线评估模型的区分度及准确度,决策曲线评估模型的临床实用价值。P<0.05为差异有统计学意义。采用R软件(4.3.0版本)完成数据统计分析。
2 结 果
2.1 患者基线特征
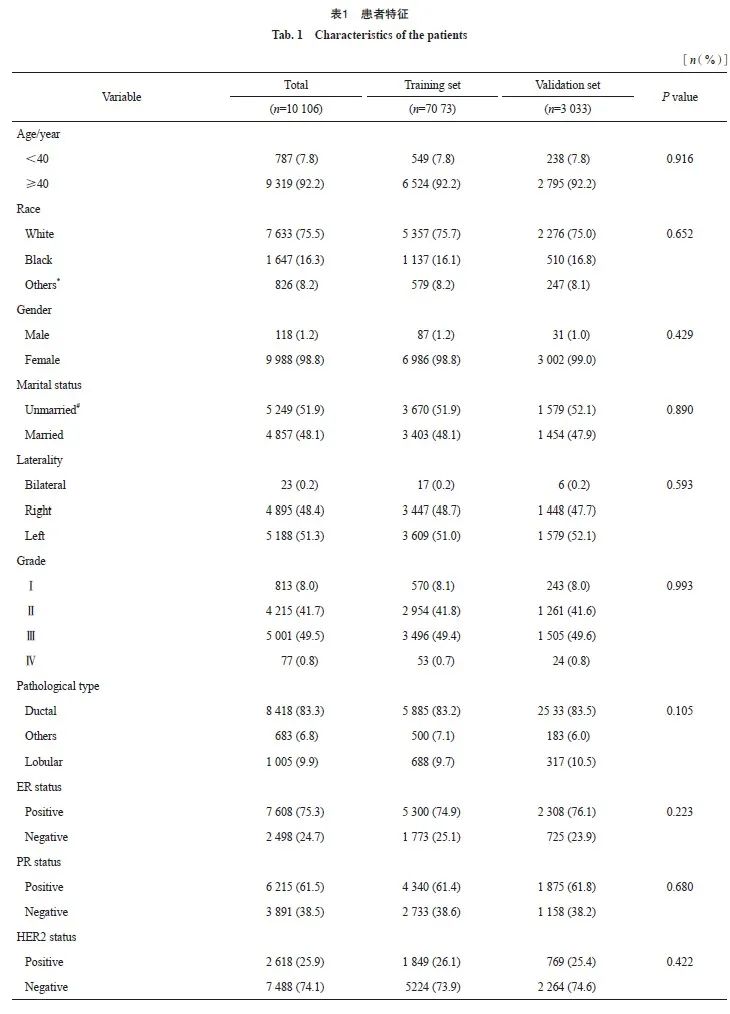
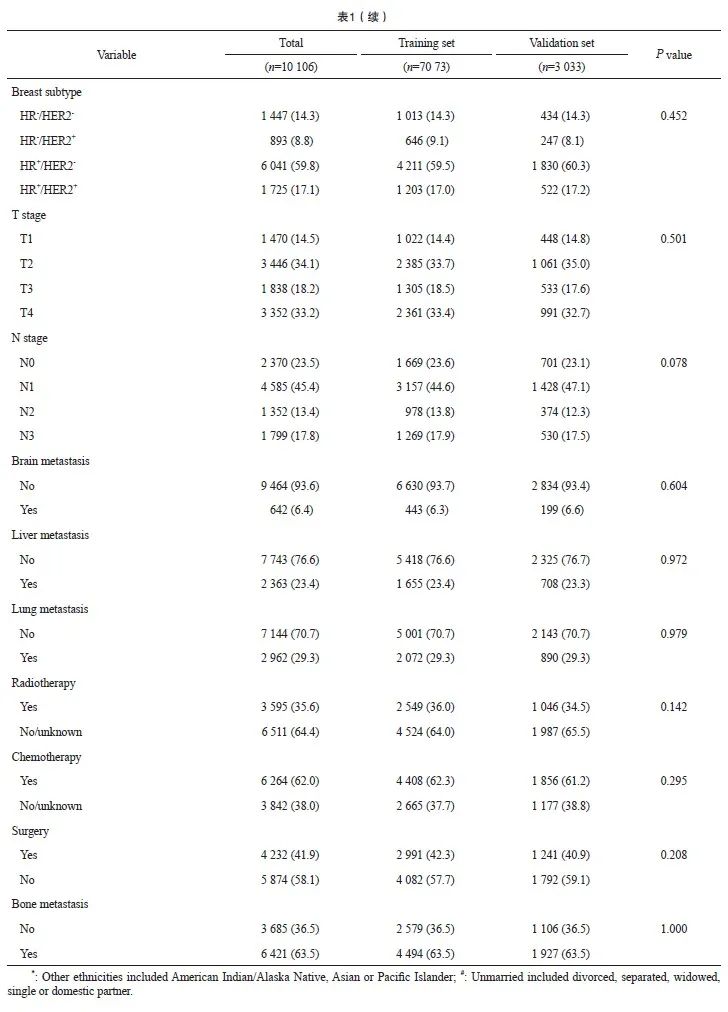
根据纳入标准和排除标准,从SEER数据库中提取10 106例转移性乳腺癌患者,随机分成训练集(7 073例)和验证集(3 033例),两组临床病理学资料差异均无统计学意义(P >0.05)。纳入患者的基线特征见表1。
2.2 乳腺癌患者发生骨转移的预测变量筛选
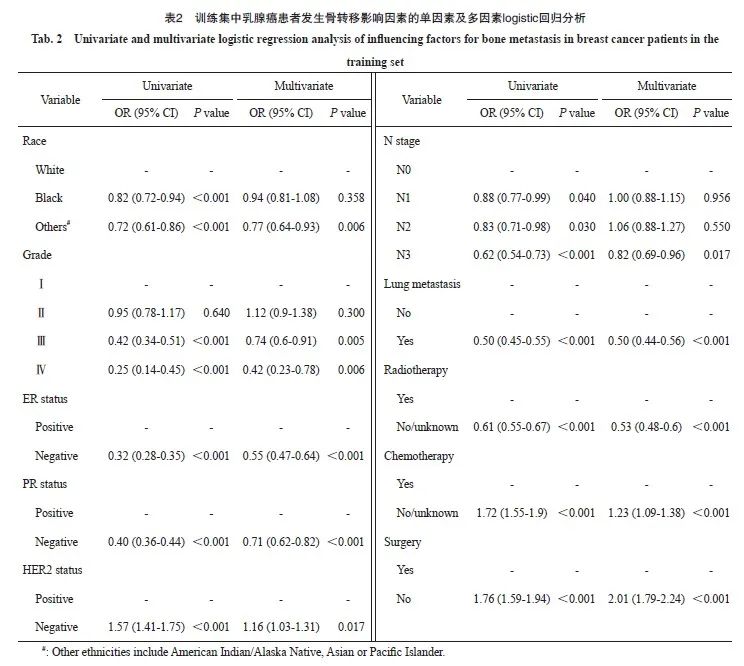
使用LASSO回归分析对训练集进行临床特征选择,19个临床变量得到10个有临床意义的变量,分别为种族、病理学分级、ER状态、PR状态、HER2状态、N分期、肺转移、放疗、化疗、手术(图1)。将上述预测变量纳入单因素和多因素logistic回归分析显示,LASSO回归分析得到的10个变量均为骨转移相关的独立因素(表2)。
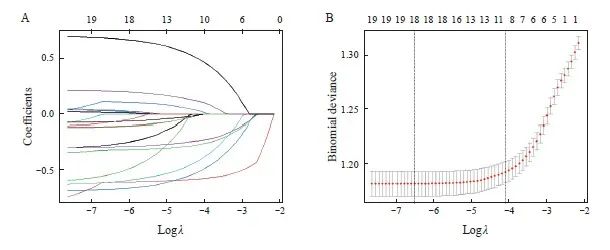
图1 LASSO回归分析进行临床特征选择
Fig. 1 LASSO regression analysis for clinical feature selection
A: Coefficient distributions of models drawn for logarithmic (lambda) sequences at different penalty levels; B:10 times cross-validation error, the first vertical line is the minimum error, the second vertical line is the cross-validation error of 1 times the minimum standard deviation.
2.3 预测模型的构建与评价
以种族、病理学分级、ER状态、PR状态、HER2状态、N分期、肺转移、放疗、化疗、手术10个变量作为预测因子,利用DT、EN、KNN、LightGBM、LR、NN、RF、SVM和Xgboost等9种机器学习算法构建模型。采用5折交叉验证的方法对各模型性能进行评估,如图2所示,神经网络和极限梯度提升算法的性能最佳。
用训练集和验证集对机器学习算法构建的预测模型进行评估。ROC及AUC分析结果表明,极限梯度提升算法构建的模型具有较好的区分能力(图3);校准曲线表明,极限梯度提升算法构建的模型具有很好的预测性能(图4);决策曲线表明,极限梯度提升算法构建的模型具有良好的临床获益性(图5)。因此综合考虑基于极限梯度提升算法构建的预测模型性能最优。
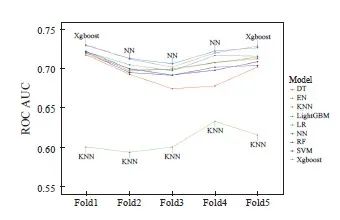
图2 机器学习算法的五折交叉验证图
Fig. 2 Five-fold cross-validation graph of a machine learning algorithm
DT: Decision tree; EN: Elastic net; KNN: K-nearest neighbor; LightGBM: Light gradient boosting machine; LR: Logistic regression; NN: Neural network; RF: Random forest; SVM: Support vector machine; Xgboost: Extreme gradient boosting.
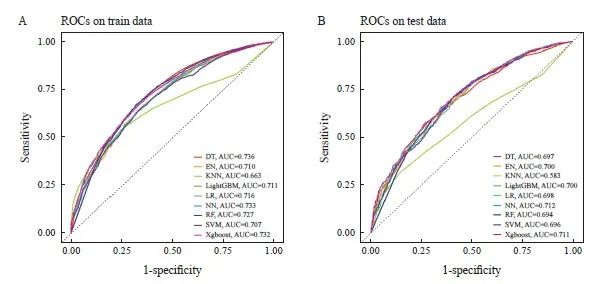
图3 机器学习算法的ROC曲线
Fig. 3 ROC curve of machine learning algorithm
A: Training set; B: Validation set.
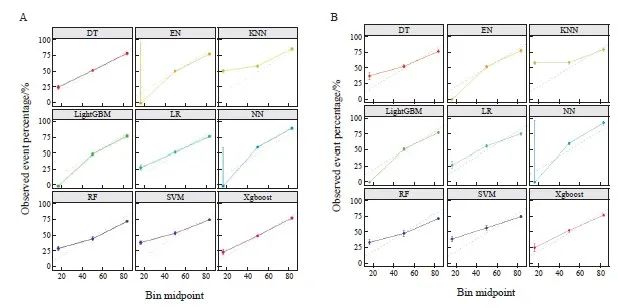
图4 机器学习算法的校准曲线
Fig. 4 Calibration curve of machine learning algorithm
A: Training set; B: Validation set.
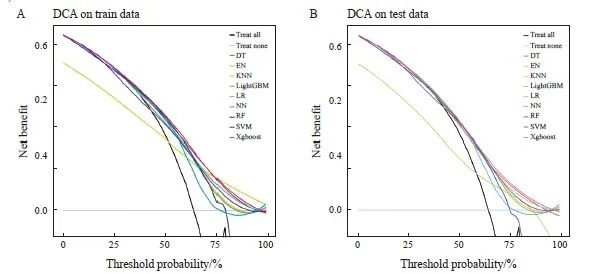
图5 机器学习算法的决策曲线
Fig. 5 Decision curve of machine learning algorithm
A: Training set; B: Validation set.
2.4 最优模型的特征重要性排名
在极限梯度提升模型中,变量差异性量化为变量重要性(图6),它可以反映每个变量对发生骨转移的贡献情况。在模型中变量的重要性按以下顺序排列:手术、放疗、肺转移、ER状态、化疗、病理学分级、PR状态、N分期、 HER2状态、种族。在所有的特征变量中,最重要、贡献度最高的是手术,这提示乳腺癌患者手术治疗情况与骨转移可能存在较为紧密的联系。
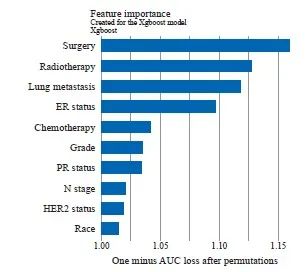
图6 极限梯度提升模型中重要特征排名
Fig. 6 Ranking of important features in the Xgboost model
2.5 基于极限梯度提升模型建立网络计算器
最后,本研究建立了一个简明的、可视化的、动态的基于极限梯度提升模型的网络计算器,可以根据患者的临床信息计算骨转移的实时风险,得到的预测值越大说明骨转移发生率越高。例如,当一个乳腺癌患者表现以下临床特征:白种人,病理学分级为Ⅱ级,ER状态阳性、PR状态阳性、HER2状态阴性、N0期、无肺转移、未接受放疗或化疗、行手术治疗。我们将以上信息输入网络计算器,然后应用集成每个因素自动计算,结果显示该患者发生骨转移的概率约为75.48%(图7)。网页计算器的链接为https://bcbm.shinyapps.io/DynNomapp/,这个网络计算器方便临床应用,并可以帮助临床医师作出更合理的诊疗决策。
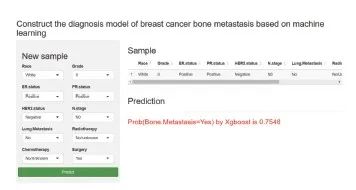
图7 预测乳腺癌患者骨转移发生概率的网络计算器
Fig. 7 A network calculator for predicting the probability of bone metastasis in breast cancer patients
2.6 模型解释
基于SHAP实现模型的可解释性。根据网络计算器输入的患者特征,绘制SHAP局部解释图,并显示各特征变量对结果影响的贡献权重(图8)。其中,N0期、行手术和放疗情况无/未知是发生骨转移的保护因素,未发生肺转移、 ER状态和PR状态阳性、HER2状态阴性、病理学分级Ⅱ级、化疗情况无/未知、白种人是发生骨转移的危险因素。
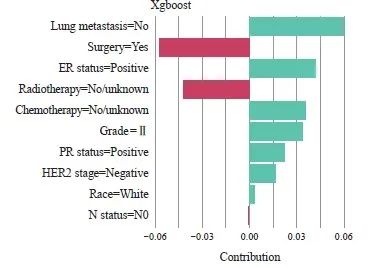
图8 SHAP局部解释图
Fig. 8 SHAP local interpretation chart
3 讨 论
乳腺癌是全球重大公共卫生问题,骨是乳腺癌远处转移最常见的部位,约占所有转移病例的70%[11]。乳腺癌骨转移可引起一系列并发症,包括剧烈疼痛、病理性骨折、高钙血症、脊髓压迫等,给患者身体活动带来极大不便,影响患者的生活质量[12]。转移性复发是乳腺癌患者死亡的主要原因[13]。尽管乳腺癌骨转移发生率很高,但乳腺癌患者并没有常规进行筛查骨转移的依据[14]。临床上,骨放射性核素显像是骨转移筛查的最常用方法,一般是患者出现相应骨痛、病理性骨折、碱性磷酸酶升高或高钙血症等可疑骨转移临床表现的筛查手段。意大利的两项随机临床试验[15-16]未能证明包括骨扫描在内的检查对乳腺癌患者发现骨转移有效。
实施早期诊断可帮助临床医师在这种困境中作出决策,开发临床预测模型筛选乳腺癌骨转移高危患者显得至关重要。机器学习技术以其强大的计算能力在医疗保健领域得到广泛应用。这项人工智能技术可以在短时间内通过分析、训练和建模大量医疗数据来预测转移性疾病的可能性,帮助诊断和评估预后[17-18]。机器学习在临床肿瘤学中被越来越多地用于诊断癌症,预测患者的临床结局,并为治疗规划提供信息。目前,已经有基于机器学习算法构建癌症预测模型。使用机器学习算法预测淋巴结转移已在甲状腺癌中得到证实[18]。Qiu等[19]基于机器学习构建直肠癌远处转移预测模型。Feng等[20]开发了预测肾癌淋巴结转移风险的机器学习模型。Xi等[21]通过机器学习和临床数据改进甲状腺癌诊断的研究。
疾病的预测因素对于患者的咨询和治疗选择的建议非常重要。为此,本研究利用机器学习建立易于使用且有效的乳腺癌骨转移预测模型,以预测乳腺癌患者发生骨转移的概率。本研究中,有三项重要结论。① 筛选出骨转移的独立危险因素,分别是种族、病理学分级、ER状态、PR状态、HER2状态、N分期、肺转移、放疗、化疗、手术。② 比较9个基于机器学习算法的模型,极限梯度提升模型具有最佳的预测性能。③ 构建基于极限梯度提升模型的网络计算器。
决定乳腺癌骨转移的潜在机制很复杂,受许多因素影响。种族是乳腺癌骨转移发生的独立危险因素[22-23]。这表明乳腺癌发生骨转移可能存在种族异质性,与不同种族的遗传、生活方式、环境因素等有关。一项研究[24]显示,原发乳腺癌的黑种人患者更易发生骨转移。本研究显示,其他人种不容易发生骨转移,这可能与研究的样本量较小有关,需要对不同种族的人群开展进一步研究。
病理学分级是乳腺癌患者发生骨转移的危险因素[25]。有研究[26]显示,在所有确诊的Ⅰ期和Ⅲ期乳腺癌患者中,13.6%的患者最终会出现骨转移。而随着病理学分级的升高,骨转移的百分比在下降[27],这表明侵袭性较低的乳腺癌与骨转移的发展相关。
本研究显示,ER、PR和HER2状态与骨转移有关。有研究[28]提示,低级别和ER阳性乳腺癌更有可能与骨转移的发展相关,其原因可能和ER阳性、肿瘤体积较大、临床分期较晚有关[29],还可能与不同信号转导通路的激活有关[30-31]。ER阳性/PR阴性乳腺癌患者的骨转移率显著升高,这表明ER阳性/PR阴性的乳腺癌亚组可能具有较高的骨转移风险[25]。而Loi等[32]的研究表明,在ER阳性的乳腺癌症患者中,PR是区分高级别和低级别恶性亚组的关键因素。这些研究提示明确乳腺癌分型有利于预测骨转移的发生情况,并可以指导乳腺癌的精准内分泌 治疗。
有研究[33]报道,乳腺癌发生骨转移与N分期有关,这与本研究结果一致。不同的原发性肿瘤有转移到不同器官的倾向,原发性肿瘤转移至不同器官的转移模式通常称为器官特异性转移[34]。根据最近的研究,骨骼和肺是乳腺癌转移最频繁的部位[35-36]。本研究发现,乳腺癌发生肺转移后再发生骨转移的风险降低。
乳腺癌的治疗涉及多学科综合治疗,包括手术、放疗和化疗[37]。本研究显示,积极的综合治疗有利于防止乳腺癌发生骨转移,这说明确诊乳腺癌后,进行规范、系统的个体化治疗尤为重要。对于可切除的乳腺癌,本研究建议尽可能进行原发部位手术,以减少骨转移的发生情况。近年来有研究[38]表明,原发性肿瘤手术也可以用作姑息治疗,以控制肿瘤负荷并让转移性乳腺癌患者生存获益。研究[39]表明,根治性乳房切除术明显优于保乳手术和部分乳房切除术,是最好的手术选择。因此,在健康条件允许、符合手术指征的情况下,本研究建议对乳腺癌患者尽可能进行原发灶手术。及时手术有助于防止肿瘤细胞向周围区域侵犯,延长患者的生存时间,提高患者的生活质量。
另外,有研究[40]显示,随着乳腺癌诊断年龄的增加,发生骨转移风险会显著降低。加拿大的一项关于女性乳腺癌骨转移的发生率、预测因素以及转移后生存率的前瞻性队列研究显示,高龄对骨转移有保护作用[26]。年轻的乳腺癌患者更加需要防范骨转移的发生。不同的乳腺癌病理学类型发生骨转移的风险也不同,浸润性小叶癌比浸润性导管癌更有可能首先发生骨转移[40]。
Xgboost是一种先进的集成机器学习算法,具有快速并行处理、高性能、可扩展到大数据以及处理稀缺数据的能力等优势[41-42]。Xgboost已经应用于各种临床医学领域,例如糖尿病的检查[43]、预测甲胎蛋白阳性肝细胞癌患者的预后[44]以及预测外周动脉疾病腹股沟下搭桥手术后的疗效[45]。本研究运用大样本数据分析乳腺癌骨转移的风险因素,构建基于极限梯度提升模型的网络计算器,便于临床医师在日常诊疗 中使用。
本研究也存在一些局限性。首先,SEER数据库中没有包括内分泌治疗和靶向治疗的使用情况,因此无法评估这些治疗对骨转移的影响。此外,我们在同一人群中对模型进行验证,这对模型性能的评估可能存在偏差。因此,本研究开发基于最优模型的网络计算器,方便临床医师使用外部数据评估模型的预测性能。
综上所述,本研究采用9种机器学习算法构建乳腺癌骨转移预测模型,采用ROC曲线、校准曲线和决策曲线评价模型性能,研究结果显示,极限梯度提升模型具有最佳的预测能力。因此,我们基于极限梯度提升模型开发预测乳腺癌骨转移的网络计算器,希望能够帮助临床医师筛查乳腺癌骨转移的高危患者,及时预测乳腺癌发生骨转移的可能性,并帮助临床医师作出更合理的临床决策。
利益冲突声明:所有作者均声明不存在利益冲突。
作者贡献声明:
欧阳飞:实施研究,分析数据,论文撰写及修改;王阳、陈瑜、裴国清:数据整理,统计学分析;王陵、张扬:研究指导,数据分析指导;石磊:课题设计,研究指导,论文修改,论文审核。
[参考文献]
[1] SUNG H, FERLAY J, SIEGEL R L, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249.
[2] MEDEIROS B, ALLAN A L. Molecular mechanisms of breast cancer metastasis to the lung: clinical and experimental perspectives[J]. Int J Mol Sci, 2019, 20(9): 2272.
[3] LIANG Y R, ZHANG H W, SONG X J, et al. Metastatic heterogeneity of breast cancer: molecular mechanism and potential therapeutic targets[J]. Semin Cancer Biol, 2020, 60: 14-27.
[4] JIANG Z C, LI J Y, CHEN S J, et al. Zoledronate and SPIO dual-targeting nanoparticles loaded with ICG for photothermal therapy of breast cancer tibial metastasis[J]. Sci Rep, 2020, 10(1): 13675.
[5] ZAJKOWSKA M, LUBOWICKA E, FIEDOROWICZ W, et al. Human plasma levels of VEGF-A, VEGF-C, VEGF-D, their soluble receptor-VEGFR-2 and applicability of these parameters as tumor markers in the diagnostics of breast cancer[J]. Pathol Oncol Res, 2019, 25(4): 1477-1486.
[6] SIDEY-GIBBONS J A M, SIDEY-GIBBONS C J. Machine learning in medicine: a practical introduction[J]. BMC Med Res Methodol, 2019, 19(1): 64.
[7] ESTEVA A, KUPREL B, NOVOA R A, et al. Dermatologistlevel classification of skin cancer with deep neural networks[J]. Nature, 2017, 542(7639): 115-118.
[8] ANDERSON J P, PARIKH J R, SHENFELD D K, et al. Reverse engineering and evaluation of prediction models for progression to type 2 diabetes: an application of machine learning using electronic health records[J]. J Diabetes Sci Technol, 2015, 10(1): 6-18.
[9] RAHIMIAN F, SALIMI-KHORSHIDI G, PAYBERAH A H, et al. Predicting the risk of emergency admission with machine learning: development and validation using linked electronic health records[J]. PLoS Med, 2018, 15(11): e1002695.
[10] GUO Q P, WANG Y Q, AN J, et al. A prognostic model for patients with gastric signet ring cell carcinoma[J]. Technol Cancer Res Treat, 2021, 20: 15330338211027912.
[11] YANG Y P, MA Y X, SHENG J, et al. A multicenter, retrospective epidemiologic survey of the clinical features and management of bone metastatic disease in China[J]. Chin J Cancer, 2016, 35: 40.
[12] PLUNKETT T A, SMITH P, RUBENS R D. Risk of complications from bone metastases in breast cancer. implications for management[J]. Eur J Cancer, 2000, 36(4): 476-482.
[13] PAREEK A, SINGH O P, YOGI V, et al. Bone metastases incidence and its correlation with hormonal and human epidermal growth factor receptor 2 neu receptors in breast cancer[J]. J Cancer Res Ther, 2019, 15(5): 971-975.
[14] Recommended breast cancer surveillance guidelines. American Society of Clinical Oncology[J]. J Clin Oncol, 1997, 15(5): 2149-2156.
[15] ROSSELLI DEL TURCO M, PALLI D, CARIDDI A, et al. Intensive diagnostic follow-up after treatment of primary breast cancer. A randomized trial. National research council project on breast cancer follow-up[J]. JAMA, 1994, 271(20): 1593-1597.
[16] Impact of follow-up testing on survival and health-related quality of life in breast cancer patients. A multicenter randomized controlled trial. The GIVIO investigators[J]. JAMA, 1994, 271(20): 1587-1592.
[17] MO X L, CHEN X J, IEONG C, et al. Early prediction of clinical response to etanercept treatment in juvenile idiopathic arthritis using machine learning[J]. Front Pharmacol, 2020, 11: 1164.
[18] ZHU J, ZHENG J X, LI L F, et al. Application of machine learning algorithms to predict central lymph node metastasis in T1-T2, non-invasive, and clinically node negative papillary thyroid carcinoma[J]. Front Med, 2021, 8: 635771.
[19] QIU B X, SHEN Z X, WU S, et al. A machine learning-based model for predicting distant metastasis in patients with rectal cancer[J]. Front Oncol, 2023, 13: 1235121.
[20] FENG X W, HONG T, LIU W C, et al. Development and validation of a machine learning model to predict the risk of lymph node metastasis in renal carcinoma[J]. Front Endocrinol, 2022, 13: 1054358.
[21] XI N M, WANG L, YANG C J. Improving the diagnosis of thyroid cancer by machine learning and clinical data[J]. Sci Rep, 2022, 12(1): 11143.
[22] SAKHUJA S, DEVEAUX A, WILSON L E, et al. Patterns of de-novo metastasis and breast cancer-specific mortality by race and molecular subtype in the SEER population-based dataset[J]. Breast Cancer Res Treat, 2021, 186(2): 509-518.
[23] GAO T Y, SHAO F. Risk factors and prognostic factors for inflammatory breast cancer with bone metastasis: a populationbased study[J]. J Orthop Surg (Hong Kong), 2021, 29(2): 23094990211000144.
[24] AKINYEMIJU T, SAKHUJA S, WATERBOR J, et al. Racial/ ethnic disparities in de novo metastases sites and survival outcomes for patients with primary breast, colorectal, and prostate cancer[J]. Cancer Med, 2018, 7(4): 1183-1193.
[25] CHEN J, ZHU S, XIE X Z, et al. Analysis of clinicopathological factors associated with bone metastasis in breast cancer[J]. J Huazhong Univ Sci Technol (Med Sci), 2013, 33(1): 122-125.
[26] LIEDE A, JERZAK K J, HERNANDEZ R K, et al. The incidence of bone metastasis after early-stage breast cancer in Canada[J]. Breast Cancer Res Treat, 2016, 156(3): 587-595.
[27] GAO C W, WANG J G, HE P S, et al. Metastatic pattern of breast cancer by histologic grade: a SEER population-based study[J]. Discov Med, 2022, 34(173): 189-197.
[28] JAMES J J, EVANS A J, PINDER S E, et al. Bone metastases from breast carcinoma: histopathological-radiological correlations and prognostic features[J]. Br J Cancer, 2003, 89(4): 660-665.
[29]ARPINO G, WEISS H, LEE A V, et al. Estrogen receptor-positive, progesterone receptor-negative breast cancer: association with growth factor receptor expression and tamoxifen resistance[J]. J Natl Cancer Inst, 2005, 97(17): 1254-1261.
[30]ARCIERO C A, GUO Y, JIANG R J, et al. ER+/HER2+ breast cancer has different metastatic patterns and better survival than ER-/HER2+ breast cancer[J]. Clin Breast Cancer, 2019, 19(4): 236-245.
[31]HAYASHI N, IWAMOTO T, QI Y, et al. Bone metastasis-related signaling pathways in breast cancers stratified by estrogen receptor status[J]. J Cancer, 2017, 8(6): 1045-1052.
[32]LOI S, HAIBE-KAINS B, DESMEDT C, et al. Definition of clinically distinct molecular subtypes in estrogen receptor-positive breast carcinomas through genomic grade[J]. J Clin Oncol, 2007, 25(10): 1239-1246.
[33]KOIZUMI M, YOSHIMOTO M, KASUMI F, et al. An open cohort study of bone metastasis incidence following surgery in breast cancer patients[J]. BMC Cancer, 2010, 10: 381.
[34]TAYYEB B, PARVIN M. Pathogenesis of breast cancer metastasis to brain: a comprehensive approach to the signaling network[J]. Mol Neurobiol, 2016, 53(1): 446-454.
[35]LU X, KANG Y B. Organotropism of breast cancer metastasis[J]. J Mammary Gland Biol Neoplasia, 2007, 12(2/3): 153-162.
[36]YATES L R, KNAPPSKOG S, WEDGE D, et al. Genomic evolution of breast cancer metastasis and relapse[J]. Cancer Cell, 2017, 32(2): 169-184.e7.
[37]中国抗癌协会乳腺癌专业委员会, 中华医学会肿瘤学分会乳腺肿瘤学组. 中国抗癌协会乳腺癌诊治指南与规范(2024年版)[J]. 中国癌症杂志, 2023, 33(12): 1092-1187. The Society of Breast Cancer China Anti-Cancer Association, Breast Oncology Group of the Oncology Branch of the Chinese Medical Association. Guidelines for breast cancer diagnosis and treatment by China Anti-cancer Association (2024 edition)[J]. China Oncol, 2023, 33(12): 1092-1187.
[38]TU Q H, HU C, ZHANG H, et al. Establishment and validation of novel clinical prognosis nomograms for luminal A breast cancer patients with bone metastasis[J]. Biomed Res Int, 2020, 2020: 1972064.
[39]GAO B, OU X L, LI M F, et al. Risk stratification system and visualized dynamic nomogram constructed for predicting diagnosis and prognosis in rare male breast cancer patients with bone metastases[J]. Front Endocrinol, 2022, 13: 1013338.
[40]PURUSHOTHAM A, SHAMIL E, CARIATI M, et al. Age at diagnosis and distant metastasis in breast cancer: a surprising inverse relationship[J]. Eur J Cancer, 2014, 50(10): 1697-1705.
[41]CHEN X, LI D W. Sequencing facility and DNA source associated patterns of virus-mappable reads in whole-genome sequencing data[J]. Genomics, 2021, 113(1 Pt 2): 1189-1198.
[42]LIU G C, CHEN X, LUAN Y H, et al. VirusPredictor: XGBoost-based software to predict virus-related sequences in human data[J]. Bioinformatics, 2024, 40(4): btae192.
[43]PALECZEK A, GROCHALA D, RYDOSZ A. Artificial breath classification using XGBoost algorithm for diabetes detection[J]. Sensors, 2021, 21(12): 4187.
[44]DONG B T, ZHANG H, DUAN Y Y, et al. Development of a machine learning-based model to predict prognosis of alpha-fetoprotein-positive hepatocellular carcinoma[J]. J Transl Med, 2024, 22(1): 455.
[45]LI B, EISENBERG N, BEATON D, et al. Using machine learning (XGBoost) to predict outcomes after infrainguinal bypass for peripheral artery disease[J]. Ann Surg, 2024, 279(4): 705-713.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言
















#乳腺癌# #骨转移# #预测模型#
1