胡政/徐锦/马晴团队《Genome Biology》:提出单细胞谱系追踪的量化评分体系,为基于临床样品的细胞谱系研究提供理论指导
2025-03-27 BioMed科技 BioMed科技 发表于陕西省
中国科学院深圳先进技术研究院与中山大学团队合作研究发现,克隆扩张强度影响线粒体谱系追踪效果。通过计算仿真和单细胞多组学分析,提出 “谱系信息评分”量化突变标记准确性,为该技术应用提供理论指导。
单细胞谱系追踪可记录细胞分裂历史,为解析发育生物学规律和疾病发生机制提供了关键性研究工具。当前主流方法依赖基因编辑,通过对外源人工条形码的定向编辑来标记细胞谱系,但该方法不适用于临床样品。线粒体DNA因其高突变率和易检测性,在人体谱系追踪中展现出巨大潜力。然而,受其多拷贝性和高度动态性影响,线粒体谱系追踪的准确性和适用性尚存不确定性,需系统评估与方法优化以提升其实用性。
北京时间3月26日,中国科学院深圳先进技术研究院合成生物学研究所胡政研究员团队、马晴研究员团队联合中山大学徐锦教授团队在Genome Biology杂志在线发表了题为Clonal expansion dictates the efficacy of mitochondrial lineage tracing in single cells的研究论文。本研究结合计算仿真模型与单细胞多组学数据分析,系统评估线粒体谱系追踪技术在不同生物学情景下的适用性。结果表明,克隆扩张强度显著影响线粒体谱系追踪效果,首次提出“谱系信息评分”(LIS),可量化线粒体DNA突变的谱系标记准确性,为线粒体谱系追踪的实际应用提供关键理论指导。

文章上线截图
团队首先利用计算仿真框架模拟了不同克隆扩张强度下线粒体DNA突变的动态演化,发现胚系线粒体异质性突变(germline mtDNA heteroplasmy)仍具克隆谱系标记能力,尤其在强克隆扩张环境(如肿瘤)中追踪准确性更高。同时,细胞内低频线粒体突变可实现深度谱系树的重构,但受限于当前单细胞测序的低覆盖度,亟需开发高深度单细胞线粒体测序技术。为量化线粒体DNA突变的克隆标记能力,团队首次提出“谱系信息评分”(LIS)指标,显著提升对有效谱系标记的鉴定及对组织克隆结构的解析能力。基于多模态单细胞数据分析,研究进一步验证了线粒体谱系追踪在强克隆扩增环境(如克隆性造血、免疫反应等)中的优越性,且高LIS值的突变表现出更高的谱系追踪准确性。
总之,本研究系统解析了线粒体DNA突变在细胞分裂过程中的动态特征,量化其在不同生物学场景下的谱系追踪能力,为线粒体谱系追踪技术的应用提供理论指导,并有望推进基于临床样品的细胞命运和克隆演化研究。
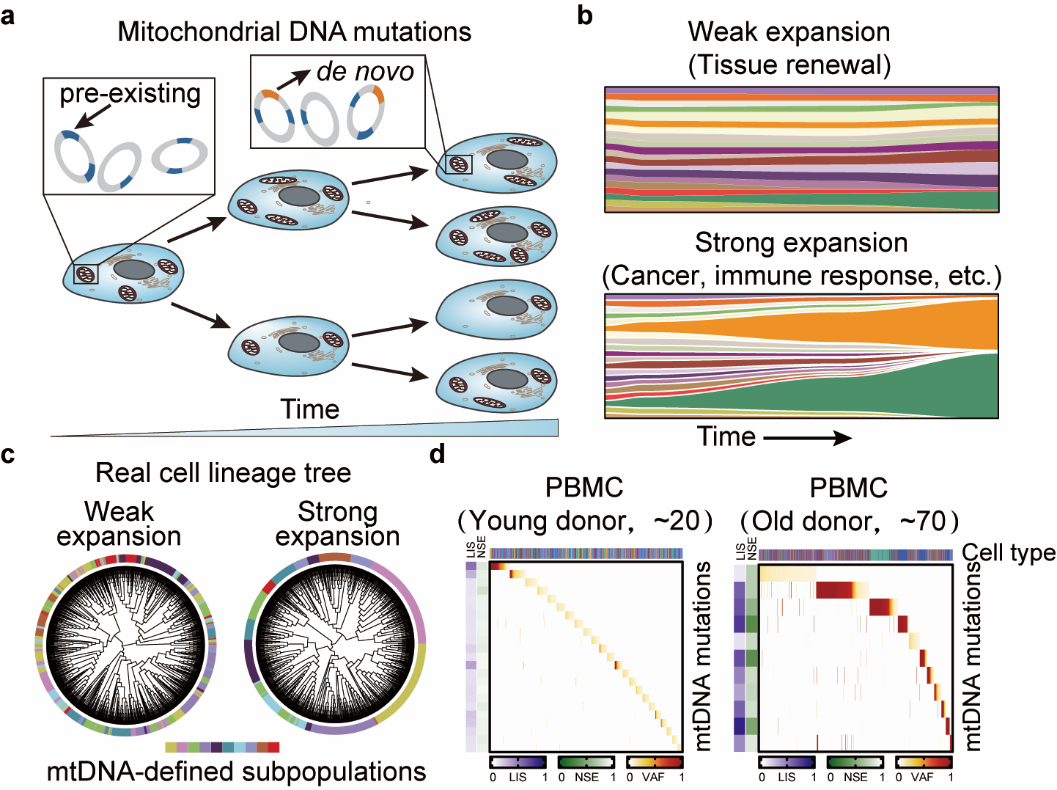
图 | 系统评估线粒体谱系追踪方法的准确性和适用性,提出单细胞谱系追踪的量化评分体系,为基于临床样品的细胞谱系研究提供理论指导。
中国科学院深圳先进技术研究院合成生物学研究所助理研究员王鑫、厦门大学数学科学学院博士研究生王琨、中山大学生命科学学院博士后张卫星为该论文的共同第一作者。中国科学院深圳先进技术研究院胡政研究员、马晴研究员和中山大学徐锦教授为该论文的共同通讯作者。该研究得到国家自然科学基金、广东省自然科学基金、深圳合成生物学创新研究院等项目的支持。
原文链接:
https://genomebiology.biomedcentral.com/articles/10.1186/s13059-025-03540-7?sessionid=-776275323
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言












#单细胞多组学# #线粒体谱系追踪#
14