DNA+RNA双组学NGS更全面检测融合基因,助力胶质瘤精准诊疗
2024-10-26 苏州绘真医学 苏州绘真医学 发表于上海
本研究开发双组学 DNA/RNA panel 用于胶质瘤综合诊断,分析 210 例病例,检测融合基因及基因突变,结果表明该 panel 对检测诊断性基因变异可靠,结合病理和分子策略有价值。
此前,我们根据 2021 年 WHO 分类系统构建了一个基于 DNA 的下一代测序(NGS)panel,用于胶质瘤的综合诊断。本研究旨在评估调整后的panel通过基于 RNA 的分析进行融合基因检测的可行性。使用这个双组学 DNA/RNA panel,我们分析了 210 例胶质瘤和其他病例,在福尔马林固定石蜡包埋组织中识别融合基因以及基因突变,包括 TERT 启动子(TERTp)突变和 1p/19q 共缺失。在 210 例患者中,有 35 例患者的肿瘤检测到融合基因。112 例胶质母细胞瘤(GB)中有 18 例携带融合基因,包括 EGFR 和 FGFR3 融合。在 IDH 突变型星形细胞瘤中,6/30 例携带融合基因,包括 MET 和 NTRK2 融合。11 例分子 GB 和 20 例未知类型病例没有基因融合。另外 11 例肿瘤,包括室管膜瘤、毛细胞型星形细胞瘤、弥漫性半球胶质瘤、婴儿型半球胶质瘤和孤立性纤维瘤检出诊断性融合基因。总体而言,本研究结果表明,根据最新的 WHO 分类,多合一双组学 DNA/RNA panel 对于检测诊断性基因变异是可靠的。病理和分子策略相结合可能在确认脑肿瘤诊断和选择治疗方案方面具有价值。
研究背景
在病理实验室中,下一代测序(NGS)越来越多地用于诊断程序。NGS panel可同时评估大量突变和拷贝数变异(CNV),进行全面基因组测序(CGP)。然而,为多种癌症类型设计的 CGP,涉及多个基因的分析,并不能完全匹配中枢神经系统(CNS)肿瘤的基因组分析要求。此外,还存在其他局限性,例如从基因组驱动的靶向治疗中获益的 CNS 肿瘤患者数量少。因此,为了扩大 CNS 肿瘤的分子靶向治疗,针对 NTRK、FGFR、BRAF、ALK、MET 和 ROS1 融合的泛癌种疗法已获批或正在临床试验中。这些治疗试验以致病性融合基因为靶点,因此,融合基因检测对于推动分子靶向精准医学在 CNS 肿瘤中的应用至关重要。由于基于 DNA 的panel分析并不总是能揭示融合基因,因此对于癌症和 CNS 肿瘤患者,需要进行全面或靶向 RNA 分析来检测融合基因,以选择分子靶向药物并扩大治疗选择。
随着 2016 年修订版世界卫生组织(WHO)分类的实施,CNS 肿瘤的诊断从基于组织学的方法转变为结合组织学和分子特征的综合诊断。在这种综合诊断策略中,检测 IDH1/2 突变、1p/19q 共缺失和 H3 K27M 突变对于诊断是必要的。诊断标准的这些变化在很大程度上导致了临床环境中脑肿瘤诊断的混乱,引发了关于如何分析分子改变以及在常规病理检查中应应用哪些方法的讨论。为了解决这些问题,我们之前构建了一个为胶质瘤定制的 DNA panel,旨在根据 WHO 分类系统,检测 1p/19q 共缺失和突变。这种综合诊断策略已经作为我院的常规分子检测应用于临床实践。更新后的 2021 年 WHO CNS肿瘤分类系统进一步修订了几项重要的诊断标准,并包括了对肿瘤分类和患者治疗的重要意义。因此,CNS肿瘤(尤其是胶质瘤)的诊断需要对其他基因变异进行分子分析,例如 TERT 启动子(TERTp)、EGFR 和 CDKN2A/B CNV、7 号染色体获得和 10 号染色体缺失(chr 7+/10−)以及融合基因。
我们先前开发的基于 DNA 的定制 panel 可以检测 TERTp 突变和 1p/19q 共缺失,本研究对该 panel 进行了调整,包括了使用基于 RNA 的测序进行融合基因分析。调整后的panel的 RNA 成分包括对 37 个代表性融合基因的分析,用于 CNS 肿瘤的分子诊断。根据最新的 WHO 分类系统,改良的双组学 DNA/RNA panel 可以同时检测综合诊断所需的诊断性基因变异。使用该 DNA/RNA 双组学 NGS panel,我们开发了一种常规临床测序策略,用于胶质瘤和其他 CNS 肿瘤治疗靶点的综合诊断和鉴定。我们使用定制的panel对 210 例 CNS 肿瘤患者的福尔马林固定和石蜡包埋(FFPE)组织进行了分析,评估作为常规分子诊断的可行性。
研究结果
病例总结
本研究纳入了 210 例患者,包括 112 例胶质母细胞瘤(GB,包括 11 例分子 GB)、30 例星形细胞瘤(AC)和 20 例未知类型(NEC)CNS 肿瘤。此外,还有 16 例少突胶质细胞瘤(ODG),7 例弥漫性中线胶质瘤(DMG),3 例弥漫性半球胶质瘤(DHG),9 例毛细胞星形细胞瘤(PA),7 例室管膜瘤,2 例节细胞胶质瘤,以及血管中心性胶质瘤、婴儿型半球胶质瘤、低级别弥漫性胶质瘤,伴MAPK 通路改变和孤立性纤维瘤(SFT)各 1 例。患者平均年龄为 55 ± 21.2 岁,有 114 例(54.3%)男性和 96 例(45.7%)女性患者。
突变和CNV
本研究使用的双组学 DNA/RNA panel 包括 51 个胶质瘤相关基因和 37 个融合基因(表 1)。在 112 例 GB 中,最常见的突变基因是 TERTp(71 %),其次是 PTEN 改变(61 %)、CDKN2A/B 纯合缺失(56 %)和 TP53 改变(38 %)。EGFR(42%)是 GB 病例中最频繁改变的酪氨酸激酶受体,其次是 PDGFRA(18%)(图 1)。在 30 例 AC 中,最常见的是 TP53 改变(90%),其次是 ATRX 改变(63%)和 PIK3CA 突变(30%)。在 3 例病例中发现了 CDKN2A/B 纯合缺失(图 2A)。在 16 例 ODG 中,PIK3CA(56%)频繁突变,其次是 FUBP1(38%)和 CIC(31%)(图 2B)。
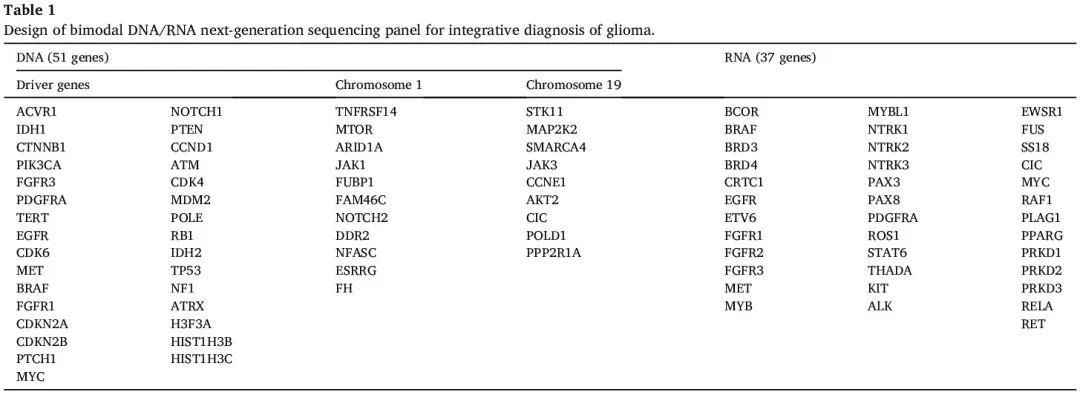
表1
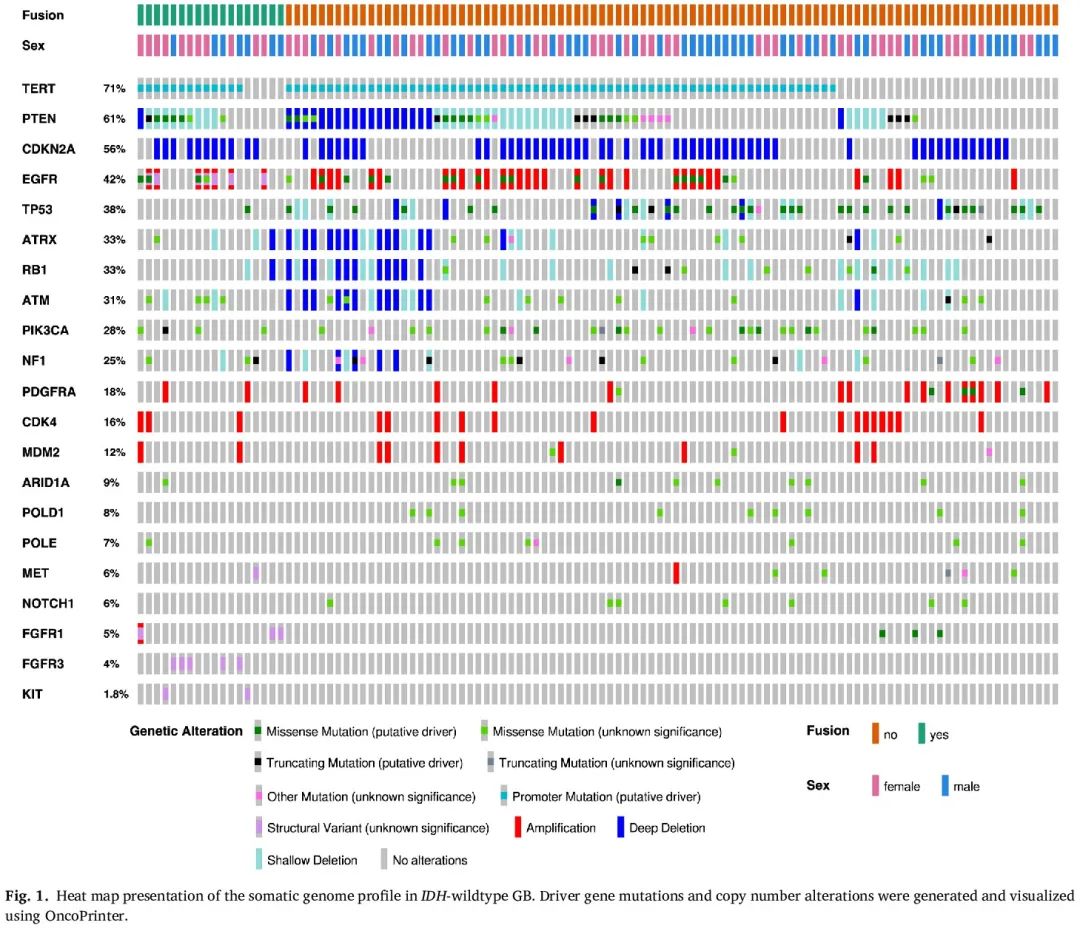
图1
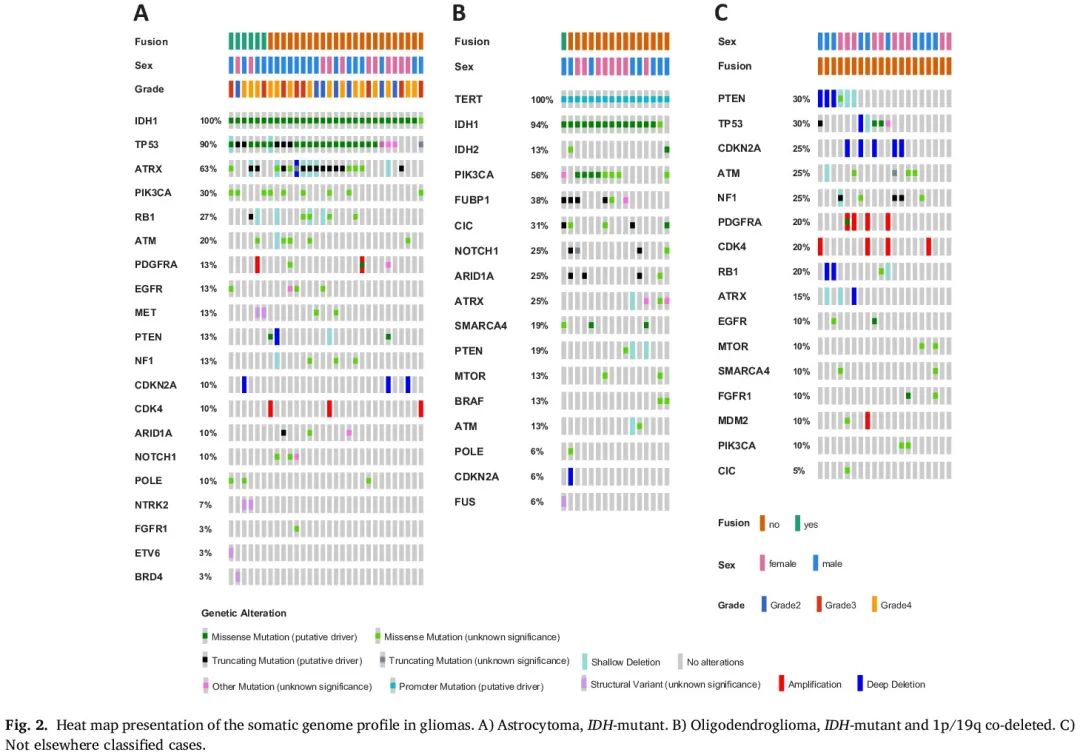
图2
分子GB和NEC病例的分子特征
根据 WHO 2021 分类系统,如果没有高级别组织病理学特征但存在以下至少一种分子特征,则诊断为 WHO 4 级 GB(分子 GB),如果不存在以下任何一项,则诊断为 NEC:TERTp 突变、EGFR 扩增或 chr 7+/10−。在本研究中,11 例为分子 GB,其余 101 例为组织学 GB。在 11 例分子 GB 中,8 例仅具有 TERTp 突变,1 例同时具有 TERTp 突变和 EGFR 扩增,1 例仅具有 EGFR 扩增,1 例仅具有 EGFR 扩增/PTEN 缺失。20 例 NEC 病例中有 4 例显示 PDGFRA 改变,是最常见的酪氨酸激酶受体改变(图 2C)。
融合基因
在 35 例(16.7%)病例中检测到融合基因,包括 NTRK1、NTRK2、FGFR1、FGFR3 和 MET 融合(表 2)。在 1 例 AC 中检测到 2 个融合基因(PTPRZ1::MET 和 ETV6::RERGL),在 1 例 GB 中检测到另外 2 个融合基因(GDF11::FGFR1 和 FGFR1::CPM)。在 1 例 GB 病例中检测到 3 个融合基因(EGFR::C1orf116、EGFR::VSTM2A 和 EGFR::FCAMR)。总体而言,融合基因在 GB 中最常见,其次是 AC(图 3A)。在分子 GB 和 NEC 病例中未检测到融合基因。
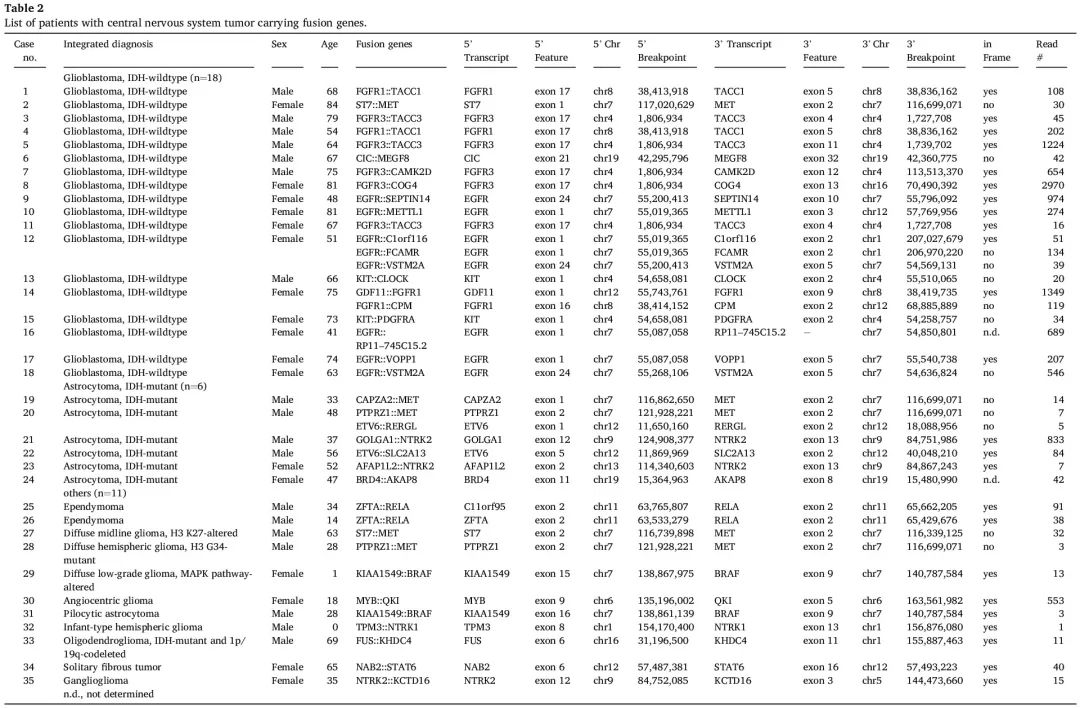
表2
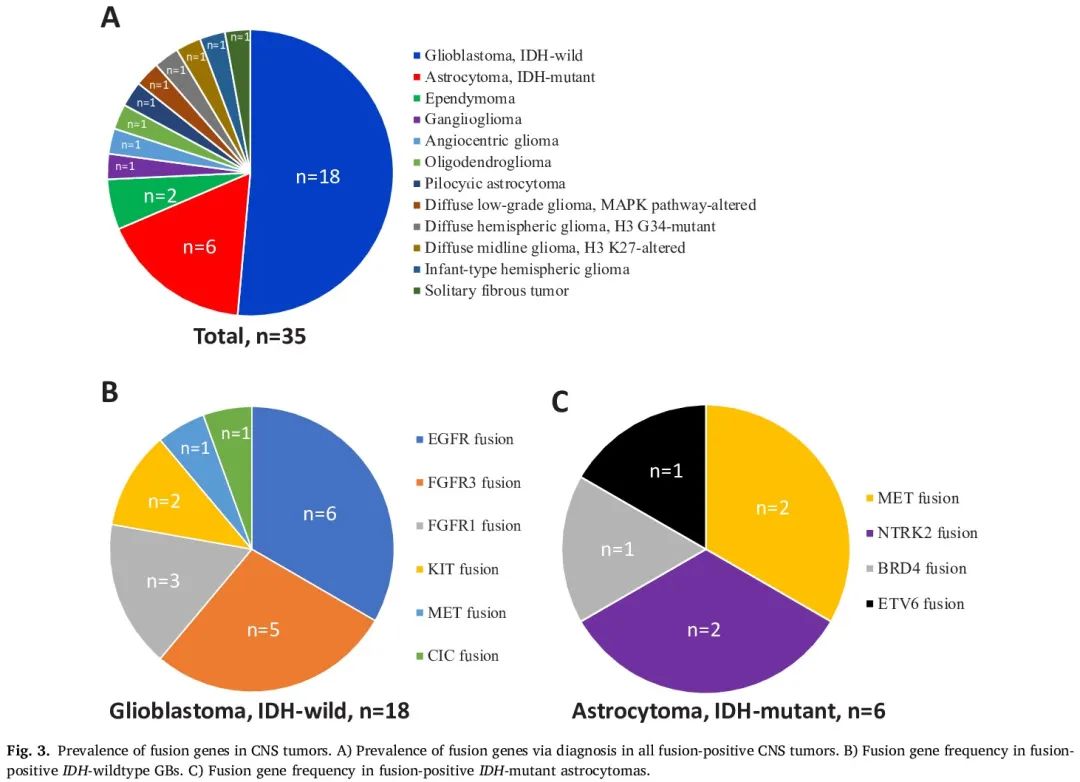
图3
在 112 例 GB 病例中,18 例患者(16.1%)检测到融合基因,而其余 94 例患者(83.9%),包括 11 例分子 GB 病例,没有融合基因。最常见的融合基因是 EGFR、FGFR3 和 FGFR1(图 3B)。有和无融合基因的患者的平均年龄和性别没有显著差异(分别为 p = 0.940 和 p = 0.305)。RB1 和 TP53 突变和/或缺失在没有融合基因的 GB 中比在有融合基因的 GB 中更常见(分别为 37.2 % vs. 11.1 %,p = 0.032 和 44.7 % vs. 5.6 %,p = 0.001)。
在 30 例 AC 病例中,6 例(20%)检测到融合基因,包括 MET 融合基因和 NTRK2 融合基因各 2 例,其次是 ETV6 和 BRD4 融合基因各 1 例(图 3C)。1 例 ODG 显示 FUS::KHDC4 融合基因。其他病例中检测到的融合基因是诊断性分子标志物,包括 ZFTA::RELA 融合基因见于 2 例室管膜瘤(病例 25 和 26);KIAA1549::BRAF 融合基因见于各 1 例弥漫性低级别胶质瘤(病例 29)和 PA(病例 31),伴MAPK 通路改变;TPM3::NTRK1 融合基因见于 1 例婴儿型半球胶质瘤(病例 32)中;MYB::QKI 融合基因见于 1 例血管中心性胶质瘤(病例 30);NAB2::STAT6 融合基因见于 1 例 SFT(病例 34)(表 2)。
3 例病例显示多个融合基因,包括 2 例 GB(病例 12 和 14)携带框内和框外融合,1 例 AC(病例 20)携带 2 个框外融合基因。其他 7 例(2、6、15、18 号GB 病例,19 号 AC 病例,27 号 DMG 病例和 28 号 DHG 病例)携带 1 个框外融合基因。作为诊断性分子标志物的融合基因都在框内(表 2)。
讨 论
本研究开发了一种双组学 DNA/RNA panel 对 FFPE 组织进行检测,用于 CNS 肿瘤的综合诊断和识别治疗靶点。我们更新了之前的 DNA panel,包括了基于 RNA 的靶向融合基因分析。该双组学 panel 可以同时分析基因突变(包括 TERTp 突变)、CNV(包括 1p/19q 共缺失)和融合基因。该panel的性能可能优于其他需要额外分子检测的融合基因分析系统,例如荧光原位杂交(FISH)、数字PCR和焦磷酸测序,来根据2021 WHO分类评估CNV和TERTp突变。因此,双组学 DNA/RNA panel 可结合常规组织病理学检查用于 CNS 肿瘤的常规临床测序。
融合基因的识别对分子诊断做出了重大贡献,改善了风险分层和新型疗法的开发。在这方面,基于 RNA 的 NGS 分析是检测基因融合的最合适方法。基于 RNA 的方法的一个优点是它涵盖了所有融合,而无需事先了解伴侣基因或断点。因此,与基于 DNA 的panel相比,基于 RNA 的方法有利于检测具有融合和外显子跳跃的表达基因。此外,通过 mRNA 剪接去除了在基于 DNA 的分析中导致技术问题的内含子区域,使基于 RNA 的方法能够更直接地捕获和/或扩增融合区域。因此,在基于 RNA 的分析中,基因的内含子结构与失败因素无关。当检测融合断点位于具有几个很大且富含重复成分的内含子的融合基因时,例如 NTRK2 和 NTRK3 融合,这一点特别重要。另一项研究表明,基于 DNA 的测序检测 NTRK2 融合基因的灵敏度为 0%,所有病例均出现假阴性结果。在本研究中, 4 个 NTRK 融合基因(1 个 NTRK1 和 3 个 NTRK2 融合基因)是使用双组学 DNA/RNA panel 的 RNA 部分检测到的。分子靶向药物在 CNS 肿瘤中的应用仍然有限;一致的融合基因检测方法对于CNS肿瘤的精准医疗是必不可少的,对于 CNS 肿瘤(包括 GB),NTRK 融合基因是 NTRK 抑制剂的分子靶标。
基于 RNA 的 panel 的一个缺点是 FFPE 组织中 RNA 的降解比 DNA 的降解快得多,可能导致目标融合基因的假阴性结果。通过严格管理分析前程序,包括标本取样、福尔马林固定和石蜡块的储存条件,可以在一定程度上减轻这种不利影响。利用新鲜或冷冻组织几乎是 RNA 保存和有效提取的绝对解决办法,但这种方法无法准确比较基因图谱和组织学。应该强调的是,组织学和分子检测的整合对于综合诊断至关重要,因此,更倾向于使用 FFPE 组织进行基因检测结合常规组织学检查。另一个缺点是难以获得用于融合基因分析的对照标本,这对于验证新开发的panel是必需的。根据全球可及的胶质瘤数据库,融合基因的检出率低于基因突变,这导致临床病例用于融合基因阳性对照存在不便。因此,我们最初使用市售的对照样本来验证 RNA panel。为了提高 RNA panel 的可靠性,需要进一步积累病例。
此外,本研究产生了显著的发现,包括在一些病例中检测到多个融合基因。在具有多个融合基因的 3 例病例中,2 例 GB 包括框内和框外融合,其中框内融合基因可能是其肿瘤发生的原因。相比之下,一例 AC 携带两个框外融合基因,提示这些融合可能没有生物学效应。在其他 7 例携带单个框外融合基因的病例中,融合基因与发病机制无关。由于在基于 RNA 的 NGS 分析中经常检测到框外融合基因,因此应仔细评估对检测到的融合基因的解读。
双组学 DNA/RNA panel分析的 DNA 成分有助于诊断和表征分子 GB 病例。在 112 例 IDH 野生型 GB 中,11 例为分子 GB,101 例为组织学 GB。在 11 例分子 GB 病例中,9 例具有 TERTp 突变,但只有 2 例携带 EGFR 扩增。本研究结果与既往研究一致,表明几乎所有分子 GB 病例都携带 TERTp 突变。另一方面,DNA 分析成分也可以对属于 NEC 类别的病例进行 WHO 分类和提取。本研究中 NEC 病例的 PDGFRA 变异频率(20%)与既往研究报道的相似,该研究显示,携带 PDGFRA 扩增的 NEC 病例显示出与 GB 类似的不良预后。然而,目前,融合基因在分子 GB 和 NEC 病例中的发生率尚未确定,本研究的分子 GB 和 NEC 病例未显示任何融合基因。可能需要进一步积累病例,使用多组学分析对更多分子 GB 和 NEC 类别进行基因检测。
2019 年,日本在国民健康保险制度下推出了 CGP,基于基因变异,更好地对癌症患者进行分类,提供更好的治疗选择。但是,也存在局限性,例如 CGP 检测不用于诊断目的,只能进行一次。使用双组学 DNA/RNA panel 的优势在于它可以以低成本多次进行,不仅可以作为综合诊断工具,还可以作为确定是否实施 CGP 的筛选工具。
综上所述,我们开发了一种双组学 DNA/RNA panel 作为胶质瘤分子诊断和治疗靶点筛查的常规策略。双组学 DNA/RNA panel 是一种可靠的方法,使用 FFPE 组织,可同时检测胶质瘤相关突变、CNV 和融合基因,并与常规组织学检查相结合。这种 panel 分析策略可以促进胶质瘤患者更准确的 CNS 肿瘤诊断和治疗选择。
参考文献:
Nayuta Higa, Toshiaki Akahane, Mari Kirishima, Hajime Yonezawa, Ryutaro Makino, Hiroyuki Uchida, Seiya Yokoyama, Tomoko Takajo, Ryosuke Otsuji, Yutaka Fujioka, Yuhei Sangatsuda, Daisuke Kuga, Hitoshi Yamahata, Nobuhiro Hata, Nobutaka Horie, Masamichi Kurosaki, Junkoh Yamamoto, Koji Yoshimoto, Akihide Tanimoto, Ryosuke Hanaya, All-in-one bimodal DNA and RNA next-generation sequencing panel for integrative diagnosis of glioma, Pathology - Research and Practice, Volume 263, 2024, 155598, ISSN 0344-0338, https://doi.org/10.1016/j.prp.2024.155598.

本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言
















#胶质瘤# #融合基因#
30