Cell:中山大学联合阿里云团队利用AI揭秘隐藏的RNA病毒“圈”
2024-11-03 测序中国 测序中国 发表于上海
中山大学与阿里云团队合作开发深度学习算法 LucaProt,可准确检测 RNA 病毒聚合酶,发现大量潜在 RNA 病毒物种和超群,提升对病毒多样性和演化认知。
RNA病毒是一类将遗传信息存储在RNA分子中的病毒,在生活中无处不在,且与人类健康密切相关,例如常见的流感病毒、新冠病毒等。截至目前,人类已经明确鉴定的病毒种类约为5000,这仅为“病毒圈”的很小一部分,主要是因为传统RNA病毒鉴定方法高度依赖与已知病毒序列的同源性比对。因此,那些缺乏同源性或同源性极低的病毒很难被发现,进而导致新病毒发现的效率较低。
近年来,人工智能(AI)相关方法,特别是深度学习算法,凭借更高的准确性、更优越的性能、对其他工具更少的依赖、灵活的模型架构等,正在对生命科学的多个领域产生重大影响。虽然已有CHEER、VirHunter、Virtifier和RNN-VirSeeker等深度学习方法能够从基因组和宏基因组数据中识别病毒,但这些方法只关注核苷酸序列本身,忽视了蛋白质序列或结构信息,从而限制了它们识别高度分化RNA病毒的能力。
近日,中山大学医学院施莽教授团队和阿里云李兆融团队合作在Cell发表了题为“Using artificial intelligence to document the hidden RNA virosphere”的研究论文,报道了新开发的一种深度学习算法LucaProt。LucaProt集成了序列和预测结构信息,能够准确检测RNA依赖性RNA聚合酶(RdRP)序列。利用该方法,研究团队共确定了161979种潜在RNA病毒物种和180个RNA病毒超群,是已知病毒种类的近30倍,大幅提升了领域内对RNA病毒多样性和病毒演化历史的认知。

主要研究内容
1 深度学习揭示RNA病毒圈的“暗物质”
研究人员共组装了10487个宏转录组。基于该数据集,研究人员使用LucaProt和ClstrSearch两种不同的策略揭示并交叉验证了潜在的病毒RdRPs,共发现了513134个RNA病毒组,代表了161979个假定的病毒物种以及180个RNA病毒超群。
随后,对该研究和其他研究中具有统一定义的RdRP蛋白序列进行自动比较,发现LucaProt新鉴定的独特病毒共70458种。值得注意的是,研究还发现了60个以前未被识别和未被充分研究的病毒群体,并且其中23个病毒超群仅被LucaProt识别。
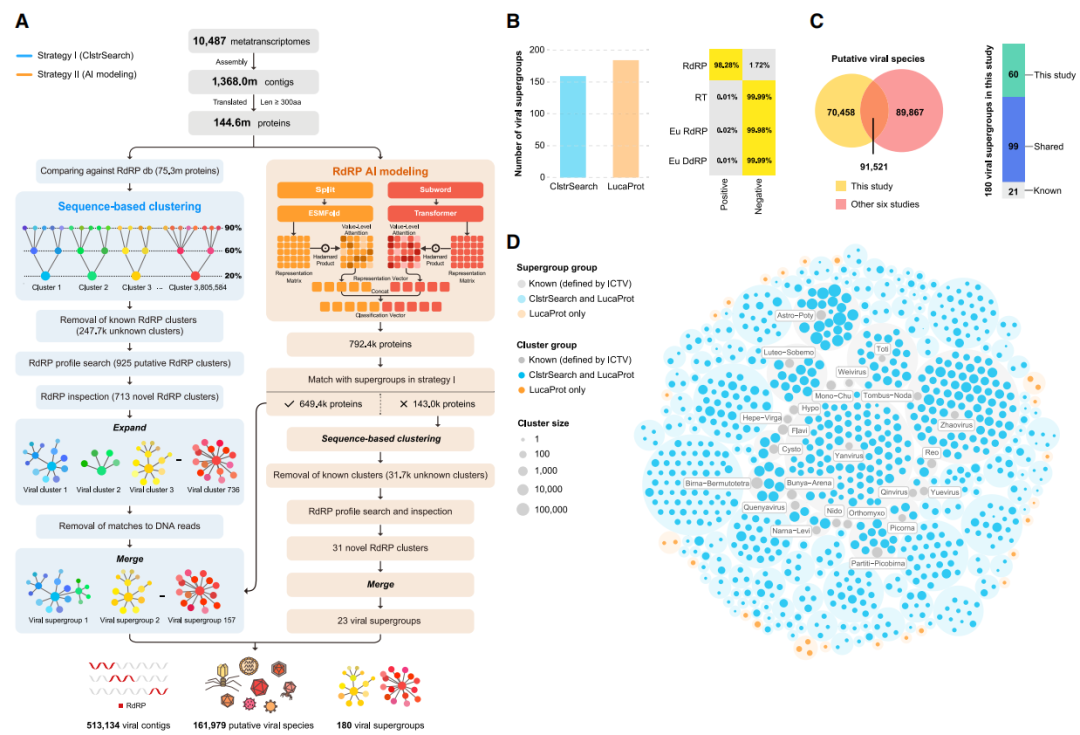
图1. 全球RNA病毒圈
为了评估LucaProt的敏感性和特异性,研究团队使用相同的数据集和RdRP数据库,并将LucaProt与其他四种病毒发现工具进行了基准测试。结果显示,LucaProt显示出最高的召回率(即正确预测的真阳性比例),同时保持相对较低的假阳性率以及合理的计算效率。在该研究中发现的所有RdRP中,LucaProt显示出最全面的病毒发现(98.22%),其他四种病毒发现工具只能识别其中一部分(76.82%-87.81%)。更重要的是,基准测试结果表明其他病毒发现工具仅识别了少数(均低于42%)LucaProt识别的新病毒。
在查全率、精确度和长序列处理方面,LucaProt也优于CHEER、VirHunter、Virtifier和RNN-VirSeeker这些RNA病毒发现工具。LucaProt还可以并行处理较长的氨基酸序列以及更好地捕获序列空间结构和部分残基之间的关系。
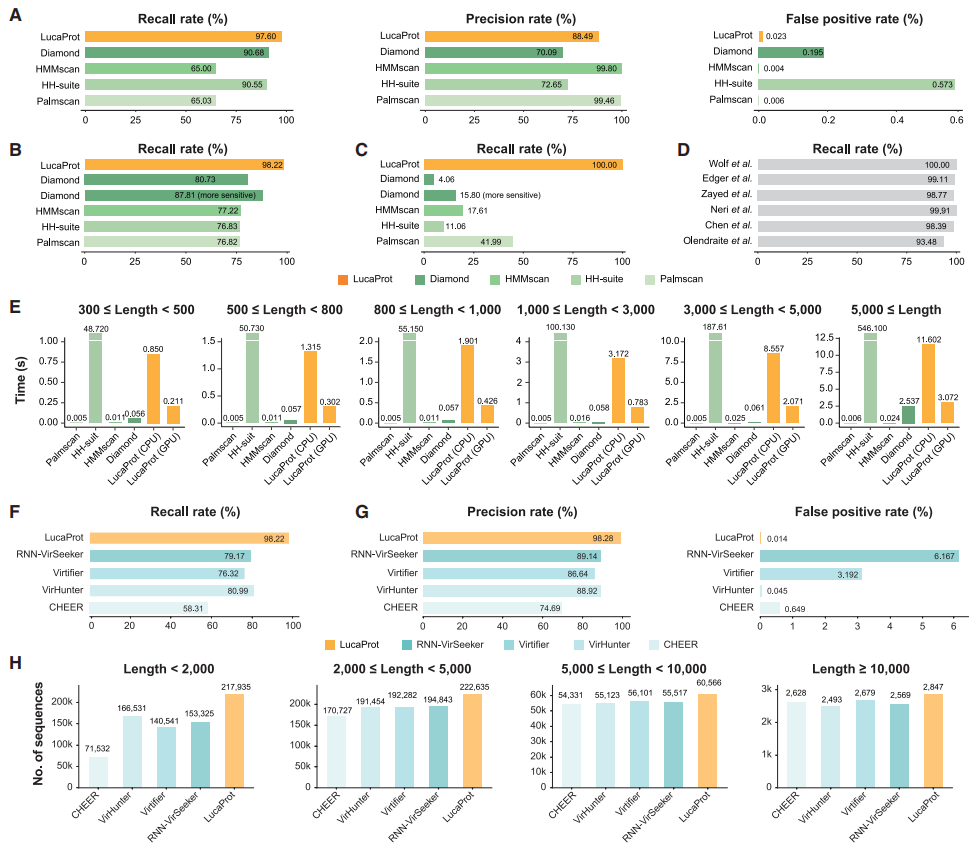
图2. LucaProt的性能评估
2 病毒超群的基因组特征
该研究鉴定的假定RNA病毒基因组的组成和结构分析显示,RdRP的基因组或基因组片段的长度在病毒超群内部间存在显著差异。此外,该数据还包含了从土壤中鉴定的较长RNA病毒基因组(47.3 kb),其属于Nido-like超群,是迄今为止鉴定的最长的RNA病毒之一。
除了RdRP外,研究团队还对新鉴定的病毒基因组编码的蛋白进行了预测和表征。虽然大多数在现有数据库中没有同源物,但仍发现了一些与已知病毒的结构蛋白(如外壳蛋白、糖蛋白)和非结构蛋白(如解旋酶、蛋白酶)相关的蛋白。在新发现的病毒超群中存在这些病毒蛋白进一步证明了它们是真正的RNA病毒。
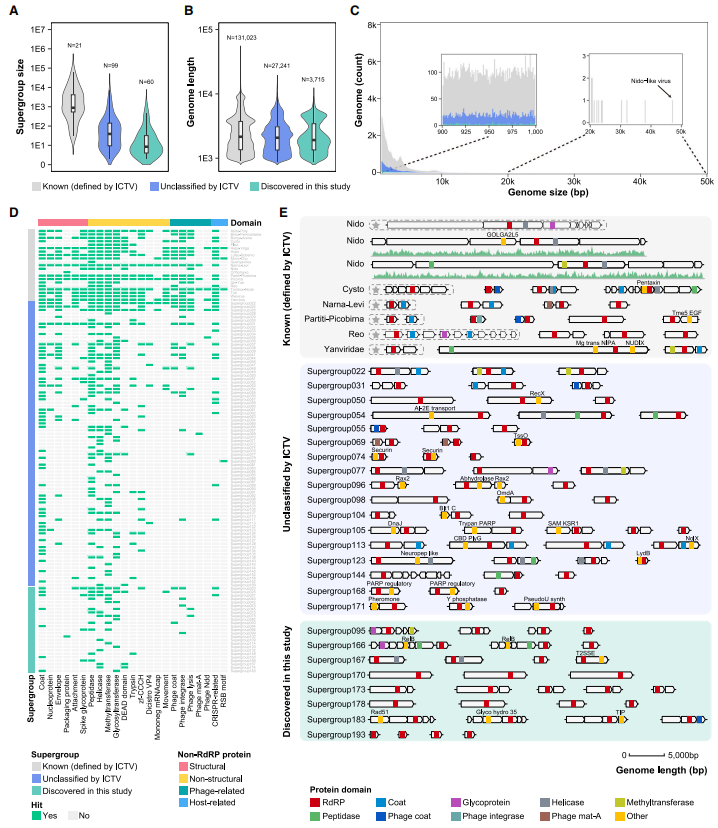
图3. 病毒超群的基因组特征
3 全球RNA病毒的生态结构
为了帮助识别不同生态模式,研究团队比较了不同生态系统亚型中RNA病毒组的α多样性和丰度水平。总的来说,平均α多样性在凋落叶、湿地、淡水和废水环境中最高,病毒丰度在南极沉积物、海洋沉积物和淡水生态系统亚型中达到峰值。相比之下,最低的平均α多样性和丰度是在岩盐和地下环境中,这也符合预期,因为它们的生物量非常低,所提供的宿主细胞也很少。
不过,在温泉和热岩浆喷口等极端生态环境中,相关RNA病毒的多样性较低,但丰度适中。值得注意的是,该研究建立的新病毒超群主要存在于水生和沉积物样本中,只有少量出现在脊椎动物和无脊椎动物样本中。需要指出的是,由于该研究分析的数据集是由不同的实验室生成,采用了不同的样本品处理、文库制备和测序程序,因此不同生态系统亚型之间病毒多样性和丰度的比较必然受到系统性偏差的影响。
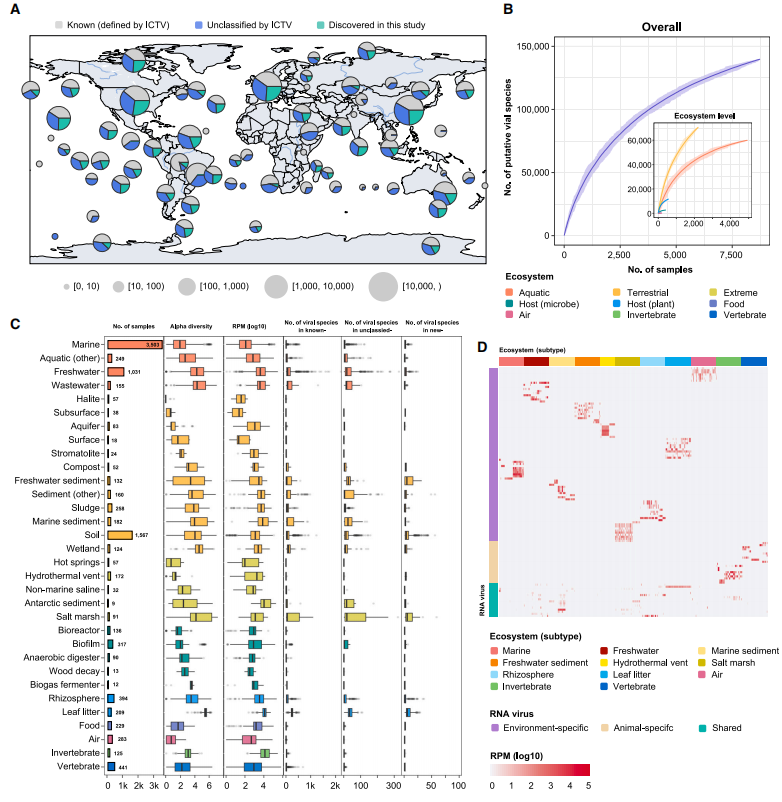
图4. 全球RNA病毒的生态结构
结 语
该研究报道了一种数据驱动的深度学习模型—LucaProt,该模型在准确性、鉴定效率和鉴定病毒多样性的广度方面优于传统方法。LucaProt不仅整合了序列数据,还囊括了结构信息,这对于准确预测蛋白质功能至关重要。总之,该研究为大规模RNA病毒发现建立了一个AI框架,一旦准备好训练数据集,就可以很容易地扩展到对任何生物“暗物质”的准确描述。
文章通讯作者施莽教授表示:“病毒的多样性远超人类想象,我们目前所看到的仍是冰山一角。AI算法模型能够挖掘出我们之前忽略或根本不知道的病毒,这种能力在疾病防控和新病原的快速识别中尤为重要。特别是在疫情暴发时,AI的速度和精度可以帮助科学家更快地锁定潜在病原体。”
文章共同第一作者、阿里云飞天实验室算法专家贺勇表示:“基于AI+病毒学的新研究框架刷新了人类对病毒圈的认识,这种认识的不断完善,有助于人类对未来可能发生的大流行进行预警,以及进一步推动RNA病毒疫苗的研发。”
论文原文:
Hou et al., Using artificial intelligence to document the hidden RNA virosphere, Cell (2024), https://doi.org/ 10.1016/j.cell.2024.09.027.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言





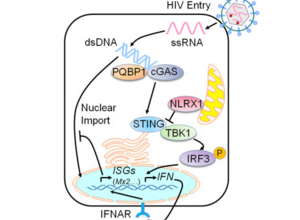








#RNA病毒# #病毒多样性#
39