Nature Methods:精准基因比对新突破:Genes2Genes框架助力单细胞轨迹分析
2024-09-21 生物探索 生物探索 发表于上海
通过对T细胞发育、IPF等模型的成功应用,G2G为细胞轨迹比对提供了强有力的工具,并展示了其在疾病机制研究、药物靶点筛选等方面的广泛应用潜力 。
引言
单细胞测序技术(single-cell RNA sequencing, scRNA-seq)近年来在生物学研究中引起了广泛关注。它能够对单个细胞内的基因表达进行精确测量,从而揭示细胞群体在时间和空间维度上的动态变化。通过这些数据,研究人员可以推断出细胞状态的转变轨迹,这在研究细胞发育、分化、疾病进展以及药物响应等动态过程中具有重要意义。传统的单细胞轨迹推断方法通常依赖于动态时间规整算法(Dynamic Time Warping, DTW),该方法能够比对两个时间序列并生成相应的匹配。然而,DTW算法存在多个局限性,尤其是对基因插入、删除(indels)等不匹配状态的处理有限,无法准确捕捉序列之间的差异。此外,现有方法还假设每个时间点必须与查询序列中的某个时间点匹配,这进一步限制了其在复杂生物过程中应用的广泛性。
针对这些局限性,9月19日Nature Methods的研究报道“Gene-level alignment of single-cell trajectories”,介绍了一种新的框架——Genes2Genes(G2G),这是一个基于贝叶斯信息论的动态规划(Dynamic Programming, DP)框架,能够在单个基因层面上对单细胞轨迹进行精确对齐。与传统方法不同,G2G允许在参考轨迹和查询轨迹之间同时捕捉匹配和不匹配的基因状态,包括基因表达的插入和删除。通过这种方式,G2G能够识别序列中关键的基因差异,尤其是那些在体外实验和体内生物系统之间表现出显著差异的基因。

单细胞测序技术的应用已经极大地推动了细胞状态转变的理解,特别是在分化和疾病模型中。然而,如何精确比对不同实验条件下的细胞轨迹仍然是一个重大挑战。现有的DTW方法无法有效处理轨迹中的不匹配问题,这限制了其在揭示关键生物学差异时的应用能力。G2G通过扩展DTW的能力,能够同时捕捉匹配和不匹配的基因状态,尤其是在复杂的生物系统中,如免疫细胞发育和疾病状态下的基因表达变化。
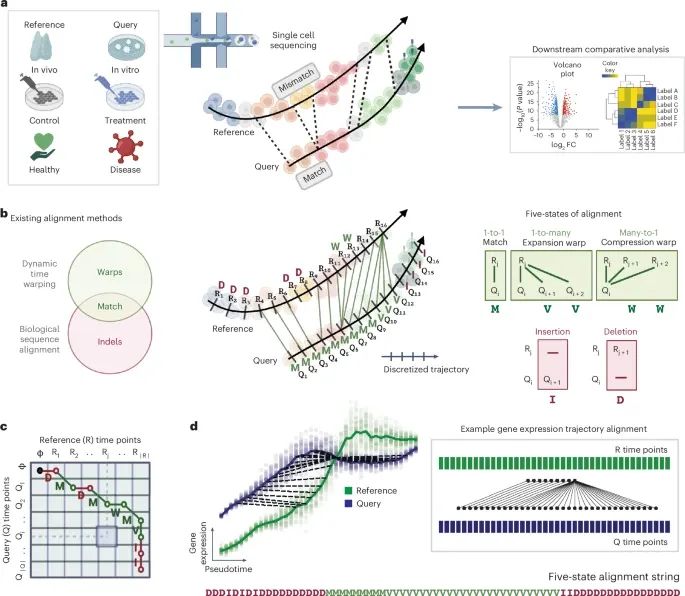
单细胞转录组轨迹比对的概念和方法(Credit: Nature Methods)
单细胞轨迹的比对原理:该图概述了将参考轨迹和查询轨迹(如体内细胞发育与体外细胞分化)进行比对的流程,展示了如何通过比对捕捉匹配和不匹配的基因表达状态。图中演示了在对照组和药物处理组、体外和体内细胞分化过程中的轨迹比对方法。
五种比对状态的理论来源:图中显示了五种比对状态,包括1对1匹配、多对一压缩、多对多扩展、插入和删除。这些状态对应不同的细胞状态变化,帮助研究人员识别出轨迹中的重要差异。
基因表达轨迹比对示例:通过对基因的轨迹进行比对,图中展示了参考和查询轨迹在伪时间上的匹配及其偏差。这包括插入、删除、扩展等不匹配情况的可视化,帮助直观了解基因表达轨迹的动态变化。
动态时间规整(DTW)与生物序列比对的结合:图中还展示了动态时间规整算法和生物序列比对在捕捉基因匹配和不匹配状态上的互补作用,进一步说明了如何通过多种方法结合来更精确地比对单细胞转录组轨迹。
G2G框架通过动态规划算法(dynamic programming),引入了贝叶斯信息论模型,可以在基因层面对细胞轨迹进行精确比对。该方法允许在参考轨迹与查询轨迹之间同时捕捉基因表达的匹配和不匹配状态,克服了传统方法无法处理插入、删除(indels)等基因变异问题的缺点。通过这种方式,G2G能够识别出不同条件下的关键基因表达差异。例如,G2G可以在体外和体内T细胞发育(Development of T cells)之间比对,揭示了体外培养的T细胞与体内发育T细胞在关键基因(如TNF信号通路(TNF signaling pathway))上的差异。
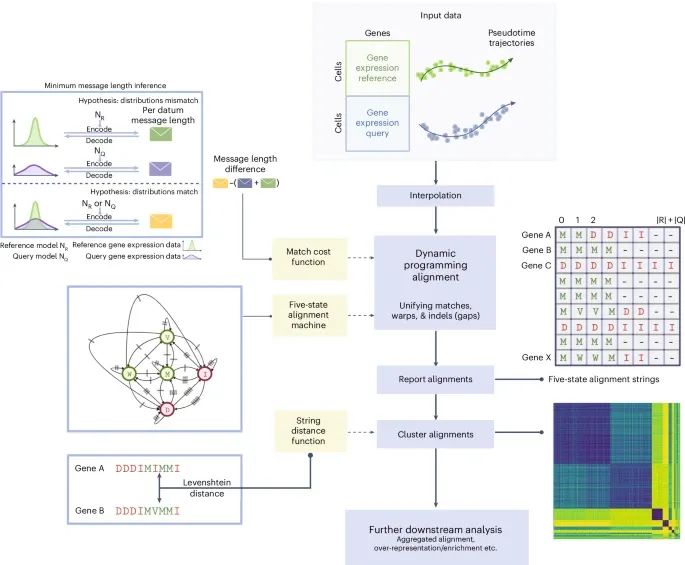
G2G框架能够在基因层面上精确对齐单细胞转录组轨迹,并支持后续的生物学分析(Credit: Nature Methods)
G2G框架的输入和预处理:图中首先展示了G2G框架的输入要求,包括参考轨迹和查询轨迹的单细胞RNA测序数据,经过log1p标准化处理后,得到细胞-基因表达矩阵,以及每个轨迹的伪时间估计值。接着,G2G会对每个基因的表达轨迹进行插值,以确保不同轨迹在相同的时间点上具有可比性。
动态规划比对算法:该图展示了G2G使用动态规划算法对基因表达轨迹进行五态比对的过程。五种比对状态包括匹配(M)、扩展(W)、压缩(V)、插入(I)和删除(D)。通过这种方式,G2G能够捕捉到参考轨迹与查询轨迹之间的复杂动态关系。
最小消息长度(MML)推断框架:图中解释了G2G使用的贝叶斯信息论模型,该模型基于最小消息长度(MML)推断来评估轨迹匹配的成本。该推断模型通过计算参考轨迹和查询轨迹在不同匹配状态下的消息长度差异,量化了两条轨迹之间的差异程度。
基因比对的聚类和统计分析:G2G不仅比对单个基因的轨迹,还对所有基因的比对结果进行聚类分析。图中展示了五态比对字符串的Levenshtein距离矩阵,通过聚类算法将具有相似比对模式的基因归类。这种聚类分析可以帮助研究人员发现具有相似动态行为的基因群,并进一步用于生物学通路分析。
下游分析的支持:G2G框架不仅能够生成基因层面的比对结果,还可以汇总所有基因的比对结果,生成细胞层面的总体比对结果。这对于理解不同轨迹中的基因群如何协同工作具有重要意义。图中展示了通过汇总分析,研究者能够进一步进行基因集的富集分析和过表达分析。
T细胞发育比对
G2G框架成功地将体外培养的T细胞与体内发育的T细胞进行比对。实验发现,体外培养的T细胞在分化过程中未能准确再现体内T细胞中的关键基因表达,尤其是在TNF信号通路中。通过G2G的比对,研究人员发现TNF信号通路中的多个基因在体外培养中被缺失,例如IL7R、KLF2、S1PR1等基因的表达较低,表明体外培养条件需要进一步优化 。
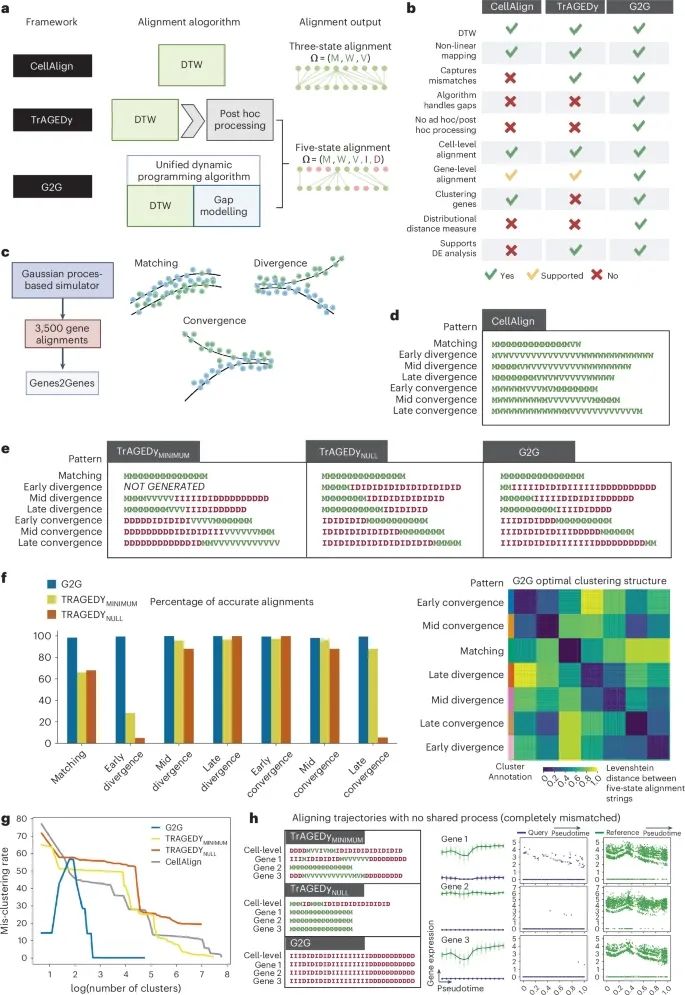
Genes2Genes (G2G) 在单细胞轨迹比对中的性能优越性,并与当前最先进的轨迹比对方法(如CellAlign和TrAGEDy)进行了对比(Credit: Nature Methods)
比对算法的差异:图中首先对比了三种不同算法的工作机制。CellAlign 仅使用了三态比对模式(匹配、扩展、压缩),而G2G和TrAGEDy 引入了五态比对模式(匹配、扩展、压缩、插入、删除)。G2G的优势在于它统一了动态时间规整算法(DTW)与插入/删除(gap)建模,能够更全面地捕捉轨迹中的不匹配状态。
模拟数据中的性能测试:在使用3500对模拟基因轨迹的测试中,图中展示了G2G在七种不同的轨迹模式下的比对表现,这些模式包括匹配、早期分歧、中期分歧、晚期分歧、早期趋同、中期趋同和晚期趋同。G2G在所有轨迹模式下的准确率都高于其他算法,尤其是在处理分歧和趋同的轨迹时,G2G的准确率接近100%。
精确度与聚类表现:图中还展示了G2G在聚类基因比对模式时的表现。在聚类时,G2G生成了准确的基因比对簇,误聚类率仅为0.1%,远远低于CellAlign的误聚类率(42.6%至60.4%)。这表明G2G不仅在单个基因的比对上表现优越,也能够准确地聚类相似的基因表达模式。
细胞层面的比对:图中展示了对两个完全不匹配的轨迹进行比对的结果。G2G成功捕捉到所有的基因不匹配情况,而TrAGEDy方法产生了部分错误的匹配段。这表明G2G在处理轨迹差异较大的情况时更加可靠。
真实数据集的应用:在真实数据集(如小鼠胰腺发育数据集)中的测试结果表明,G2G能够准确捕捉不同条件下基因表达的轨迹差异,并且相比其他方法具有更好的匹配精度和更低的误差。
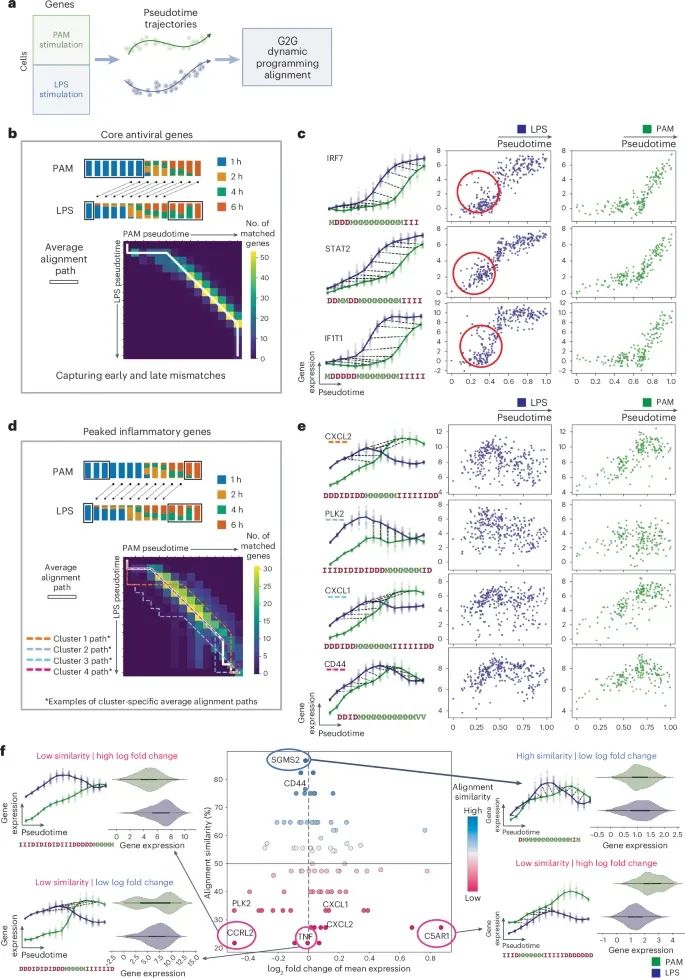
G2G框架在基因层面对单细胞轨迹进行精确比对的能力,能够有效捕捉不同条件下基因表达的匹配和不匹配状态,并为后续的基因差异分析提供了可靠的数据支持(Credit: Nature Methods)
G2G在时间序列数据中的应用:图中首先展示了G2G在一个已发表的小鼠骨髓来源的树突状细胞数据集中的应用。该数据集包含了PAM(参考)和LPS(查询)两种刺激条件下的细胞轨迹。G2G的比对结果展示了基因在两种条件下的匹配和不匹配状态,特别是发现了在早期和晚期伪时间点上的基因表达差异。
核心抗病毒基因模块的比对:图中显示了G2G对99个核心抗病毒基因进行聚合比对的结果。比对显示了PAM刺激的细胞在早期伪时间点的基因表达普遍较低,而LPS刺激的细胞则表现出较高的表达,这些差异进一步在具体基因(如IRF7、STAT2、IFIT1)的表达图中得到体现,展示了基因在不同条件下的早期表达变化。这些基因被称为“早期表达者”,它们在LPS条件下表现出提前的表达峰值。
炎症基因模块的聚类:G2G还对89个峰值炎症基因进行了聚类分析。图中展示了不同聚类簇的平均比对路径,显示出各簇基因的匹配和不匹配特征。例如,不同簇的代表基因(如CXCL2、PLK2、CXCL1和CD44)在比对路径中表现出细微的差异,有些基因在早期伪时间点表现出匹配,而另一些基因则在晚期匹配。
基因动态表达与差异表达分析:通过分析基因的比对相似性与log2倍数变化,图中展示了G2G如何通过比对发现差异表达基因。这些基因在标准差异表达分析中可能无法检测到,但通过G2G的轨迹比对,可以识别出基因在轨迹上的不匹配状态。例如,基因TNF在PAM和LPS刺激的细胞轨迹中表现出显著的不匹配,尽管其log2倍数变化较小,这表明G2G能够检测到传统方法难以捕捉的差异。
高分辨率比对:G2G在基因层面对每个时间点的匹配和不匹配进行了详细的展示。图中用黑色虚线显示了参考轨迹和查询轨迹之间的时间点匹配关系,同时展示了不同条件下基因表达的具体趋势。例如,CXCL2和TNF等基因在LPS刺激下的表现比PAM刺激更为剧烈,表现出显著的不匹配状态。
特发性肺纤维化(IPF)
在IPF的研究中,G2G比对健康与疾病患者的肺泡II型细胞(AT2 cells),揭示了在疾病早期和晚期轨迹中的基因表达差异。研究显示,IPF患者的细胞在向ABC细胞分化时,许多关键基因的表达模式与健康人存在显著差异。例如,G2G识别出AXIN2、FGFR2、ID2等基因在IPF患者中表达异常,表明这些基因可能是早期疾病进展的关键调控因子 。
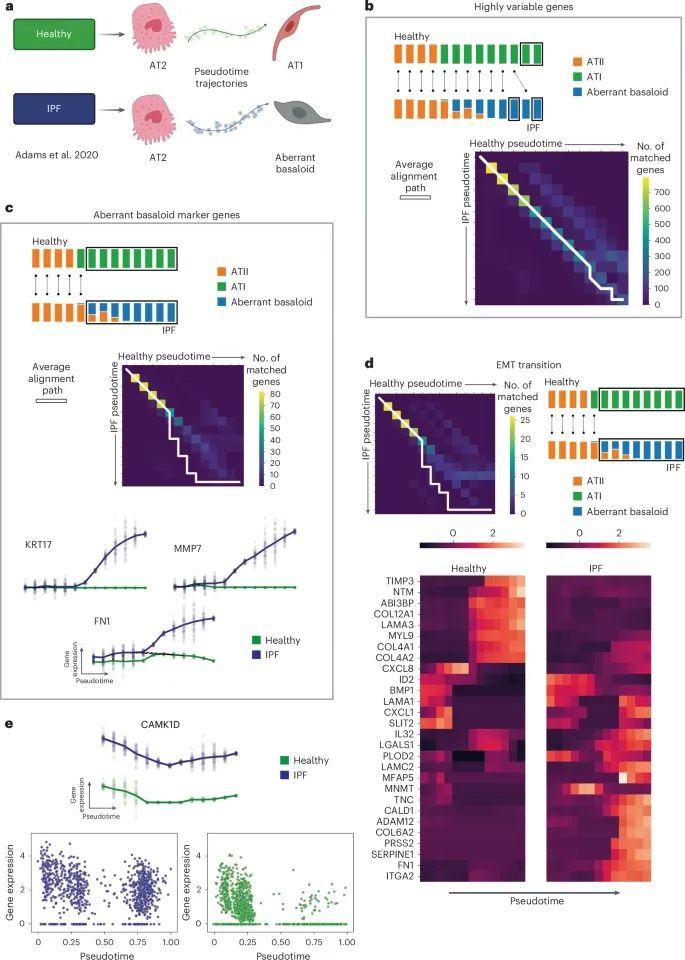
通过G2G框架比对了健康肺和IPF患者肺组织的细胞分化轨迹,揭示了多种基因在疾病状态下的表达差异,尤其是在上皮-间质转化和肺泡细胞分化相关的基因中(Credit: Nature Methods)
健康肺与IPF肺细胞分化轨迹的整体比较:图中展示了通过G2G比对健康肺与IPF肺中的细胞分化轨迹,特别是对肺泡II型细胞(AT2 cells)分化轨迹的分析。健康个体的肺细胞分化轨迹表现为较为平稳的路径,而IPF患者的肺细胞轨迹则表现出更多的分支和分歧,反映了疾病状态下细胞分化的不稳定性和复杂性。
关键基因的表达差异:通过G2G比对,研究人员发现了健康与IPF肺组织中的多个关键基因表达差异。特别是与上皮-间质转化(Epithelial-Mesenchymal Transition, EMT)相关的基因在IPF患者的轨迹中表现出显著的上调。例如,基因AXIN2、ID2、FGFR2在IPF患者中表达水平明显升高,这些基因被认为在肺纤维化的发展过程中扮演了重要角色,提示这些基因可能是疾病早期和晚期的关键调控因子。
疾病早期和晚期的细胞轨迹差异:图中还展示了IPF患者在疾病早期和晚期的细胞轨迹分化模式。通过比对,可以看到早期阶段的细胞轨迹表现出更为一致的分化路径,而晚期阶段则表现出更多的轨迹分歧和异常分化。这些轨迹异常可能与疾病进展相关,表明在疾病的不同阶段,细胞的基因表达动态发生了显著变化。
基因匹配和不匹配的细节展示:图中还通过基因比对的路径图展示了健康与IPF肺组织中具体基因的匹配和不匹配状态。例如,FOXP2、SFTPC等与肺泡细胞分化相关的基因在健康肺和IPF肺中的表达轨迹明显不同,G2G能够精确捕捉这些基因的表达不匹配状态,揭示了其在疾病状态下的异常调控。
细胞状态的聚类分析:图中通过聚类分析,将不同细胞状态的基因比对结果进行分类。结果显示,IPF患者的细胞轨迹中出现了多个异常的细胞状态簇,这些簇在健康组织中并不存在,进一步证明了IPF引起的细胞分化异常。G2G能够精确识别这些异常状态,帮助研究人员更好地理解疾病的细胞病理机制。
基因表达模式差异
G2G还揭示了体外培养与体内发育的T细胞在不同基因上的表达差异。研究发现,体内T细胞的SOX4、FOXP1、ARID5B等基因在后期发育阶段显著上调,而体外培养的T细胞未能表现出这些变化。进一步实验验证了在体外培养的T细胞中加入TNF信号能够显著提升T细胞的成熟度,这为优化体外培养条件提供了重要参考 。
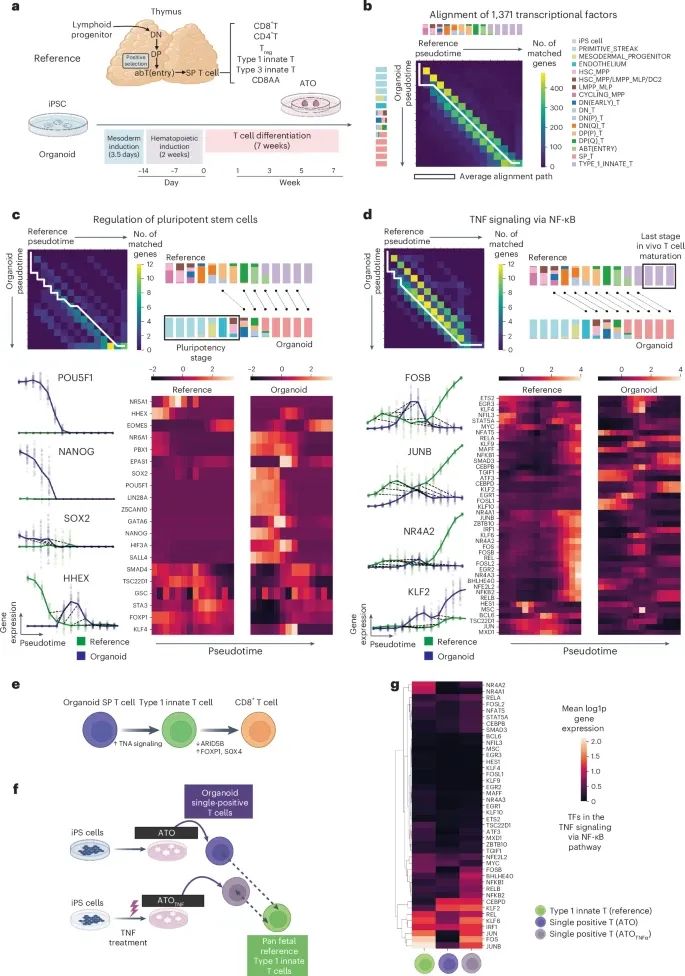
通过G2G框架比对体内和体外人类T细胞发育过程的结果,揭示了体外培养的T细胞在关键基因表达和轨迹分化上的差异,尤其是在后期阶段未能完全再现体内T细胞的发育模式(Credit: Nature Methods)
体内与体外T细胞发育轨迹的比对:图中展示了体内与体外人类T细胞发育过程的整体轨迹比对。通过G2G框架,研究人员能够比较这两种环境下T细胞分化的基因表达轨迹。结果显示,虽然体外培养的T细胞在某些阶段与体内发育的T细胞轨迹匹配,但在分化的后期阶段,体外培养的T细胞未能完全再现体内发育中的基因表达模式,尤其是在关键基因表达上存在显著差异。
关键基因表达差异的识别:通过比对,G2G框架揭示了体外T细胞发育过程中的多个关键基因未能在后期阶段表达。例如,体外培养的T细胞在后期未能表达TNF信号通路中的多个基因,如KLF2、S1PR1、IL7R。这些基因在体内发育的T细胞中是至关重要的,这表明体外培养条件在某些方面未能充分支持T细胞的成熟和功能化。
轨迹的分歧与趋同:图中展示了在不同阶段体内与体外T细胞分化轨迹的分歧和趋同状态。尤其是在早期阶段,体外T细胞的基因表达轨迹与体内较为一致,但随着分化的进行,体外T细胞的基因表达逐渐偏离体内轨迹,导致晚期阶段出现明显的分歧。这些分歧反映了体外培养条件在模拟体内微环境方面存在局限性。
体外T细胞培养条件的优化潜力:基于G2G的比对结果,研究人员通过分析体内发育的T细胞轨迹,提出了体外培养条件的优化方向。特别是,通过在培养过程中引入TNF信号,有望改善T细胞在后期的成熟度和功能表现。这一发现为进一步优化体外T细胞培养条件提供了重要的思路。
细胞状态和基因比对的可视化:图中还展示了多个基因的具体比对轨迹,特别是FOXP1、SOX4、ARID5B等关键基因在体内和体外T细胞发育中的表达差异。这些基因在体内T细胞的晚期发育中表现出显著的上调,而体外培养的T细胞未能完全再现这些表达模式。
疾病模型应用
在疾病轨迹分析中,G2G展示了其在捕捉早期和晚期基因表达差异方面的能力。例如,在IPF患者的细胞比对中,G2G识别出早期分化阶段的关键基因,揭示了这些基因在疾病进展中的潜在作用。这一发现有助于开发新的治疗靶点 。
G2G框架不仅适用于T细胞发育和IPF的研究,还展示了其在多种疾病模型中的应用潜力。特别是针对复杂的生物过程,G2G能够精确识别基因表达的匹配与不匹配状态,帮助研究人员更好地理解细胞轨迹中的关键变化。未来,随着更多数据的积累,G2G有望在细胞分化、发育及疾病进展的研究中得到更广泛的应用。
G2G框架通过其创新的动态规划算法,在基因层面实现了精确的轨迹比对。与现有的方法相比,G2G展示了更高的准确性和适应性,尤其在处理复杂的基因表达模式时表现出较强的优势。通过对T细胞发育、IPF等模型的成功应用,G2G为细胞轨迹比对提供了强有力的工具,并展示了其在疾病机制研究、药物靶点筛选等方面的广泛应用潜力 。
参考文献
Sumanaweera D, Suo C, Cujba AM, Muraro D, Dann E, Polanski K, Steemers AS, Lee W, Oliver AJ, Park JE, Meyer KB, Dumitrascu B, Teichmann SA. Gene-level alignment of single-cell trajectories. Nat Methods. 2024 Sep 19. doi: 10.1038/s41592-024-02378-4. Epub ahead of print. PMID: 39300283.
https://www.nature.com/articles/s41592-024-02378-4
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言








#单细胞测序技术# #Genes2Genes# #单细胞轨迹#
47