European Radiology:使用两阶段方法对T2加权MR图像进行多类语义和实例分割的全脊柱自动分割
2024-11-13 shaosai MedSci原创 发表于上海
机器学习是解决语义或实例分割问题的成熟工具。然而,目前还没有对整个脊柱进行分割的MRI图像的自动方法,包括脊椎骨棘突等后部元素。
MRI在临床中常用来评估脊柱,为椎间盘退变、椎体病理和椎管/脊髓结构提供有诊断价值的数据。分割技术在计算机断层扫描(CT)等成像技术中已经得到了很好的应用。在脊柱MRI中,分割不太常见,主要是由于缺乏数据、复杂的解剖结构以及后路元素的可视性差。
然而,MRI中的整个脊柱分割可以自动提取生物标志物,通过Pfirrmann分级自动检测IVD变性等退行性疾病或量化肿瘤负荷。此外,还可以进行定量分析,如确定脊柱侧凸的程度。因此,如果在临床常规中最常用的MRI序列之一(如T2w矢状涡轮自旋回波(TSE))中提供全脊柱分割,则可以提高放射学评估的质量并减少工作量。
机器学习是解决语义或实例分割问题的成熟工具。然而,目前还没有对整个脊柱进行分割的MRI图像的自动方法,包括脊椎骨棘突等后部元素。现有的T2w图像分割方法大多局限于腰椎区域,因此不能分割整个脊柱。这主要是由于与CT相比,MRI具有较低的分辨率平面,并且诸如后置元素之类的结构更难以区分。这给在MRI上执行手动注释的放射科医生带来了巨大的工作量。
相比之下,CT成像的分割已经很完善了。为了克服人工标注的问题,Graf等学者成功地使用图像翻译从MR图像创建人工CT图像。他们使用现有的CT分割模型为MRI创建分割掩模,并证明这种转换足够好地将CT级别的分割掩模转移到MRI中。

最近,发表在European Radiology 上的一篇文章提出了一种用于配对分割的脊柱相位成像网络(SPINEPS),可用于分割T2w矢状图像中颈椎、胸椎和腰椎区域的14个脊柱结构,包括语义和实例,以演示如何将自动分割模型的注释和MR-to-CT图像翻译技术相结合,同时公开一个预先训练的模型,使研究人员能够为他们的MR数据集生成脊柱分割掩码。
本项研究使用了一个公共数据集(训练/测试179/39名受试者,137名女性),一个德国国家队列(NAKO)子集(训练/测试1412/65名受试者,平均年龄53岁,694名女性)和一个内部数据集(测试10名受试者,平均年龄70岁,5名女性)。SPINEPS是一个语义分割模型,其次是滑动窗口方法,利用第二个模型从语义掩码创建实例掩码。分割评价指标包括Dice评分和平均对称表面距离(ASSD),并采用Wilcoxon符号秩检验评估统计学显著性。
在公共数据集上,SPINEPS在每个结构和度量上都优于nnUNet基线(例如,在椎骨实例上的平均值:dice 0.933 vs 0.911, p < 0.001, ASSD 0.21 vs 0.435, p < 0.001)。在NAKO的自动注释上训练的SPINEPS在合并的NAKO和内部测试分割上获得了0.918的平均全局Dice分数。添加来自公共数据集的训练数据优于此(椎骨子结构上的平均实例Dice得分为0.803 vs 0.778,平均全局Dice得分为0.931 vs 0.918)。
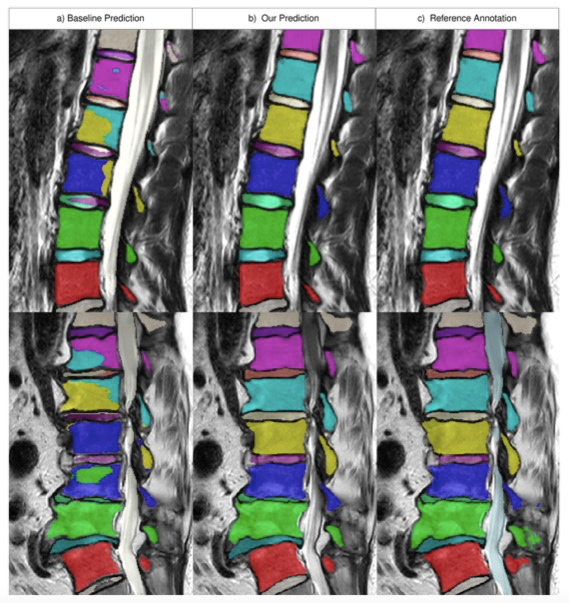
图 来自SPIDER测试集的示例。基线(a)产生典型错误的示例主题:混合不同的实例标签。我们的方法(b)非常接近参考注释(c)。我们的方法没有发生基线所产生的这种类型的错误
本项研究表明,SPINEPS可在T2w矢状面图像上对14个脊柱结构进行了分割,为临床提供了一个语义掩码和一个实例掩码来分离椎骨和椎间盘。
原始出处:
Hendrik Möller,Robert Graf,Joachim Schmitt,et al.SPINEPS-automatic whole spine segmentation of T2-weighted MR images using a two-phase approach to multi-class semantic and instance segmentation.DOI:10.1007/s00330-024-11155-y
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言









#T2# #全脊柱自动分割#
26